Introducción
El año 2020 pasará a la historia por diversos aspectos que han afectado el devenir humano, especialmente por la aparición del síndrome respiratorio agudo severo por coronavirus 2 o SARS-CoV-2 (severe acute respiratory syndrome coronavirus 2), responsable de la enfermedad infecciosa por coronavirus 2019 (COVID-19, coronavirus disease 2019). Esta enfermedad constituye la quinta pandemia con registro histórico desde la gripe española de 1918, y fue observada por primera vez como una neumonía humana atípica en habitantes de la ciudad de Wuhan, provincia de Hubei, China (Huang et al., 2020), razón por lo cual esta enfermedad se denominó inicialmente como “neumonía de Wuhan”. Con mucha probabilidad, la fecha más temprana de aparición de los síntomas del COVID-19 fue el 1 diciembre de 2019, en pacientes que presentaron un cuadro clínico inicial que incluía fiebre, malestar general, tos seca y disnea, con diagnóstico de neumonía viral (Zhu et al., 2020). Posteriormente, fueron observados otros síntomas y no fue sino hasta el 11 de enero de 2020 que el gobierno chino confirmó el primer fallecimiento por la enfermedad, un hombre de 61 años, expuesto al virus en el mercado de mariscos de Wuhan, que falleció dos días antes, el 9 de enero de 2020, después de experimentar una insuficiencia respiratoria a raíz una neumonía severa (Chen et al, 2020a).
Los resultados de la secuenciación genética demostraron sin lugar a dudas que se trataba de un nuevo virus, el séptimo miembro de la familia de los coronavirus que ha afectado a la población humana (Wu et al., 2020a) y, por esta razón el 12 de enero de 2020 la Organización Mundial de la Salud (OMS) le confirió el estatus de “nuevo coronavirus 2019” y lo denominó 2019-nCoV (WHO, 2020a). El virus fue también denominado por algunos grupos de investigación simplemente como HCoV-19 (human coronavirus 2019) (Jiang et al., 2020), sin embargo, a partir del 12 de febrero de 2020, y con el reporte de situación N° 23, la OMS comenzó a utilizar las siglas COVID-2019 (WHO, 2020b), un acrónimo que entró a formar parte del leguaje de la población, y muy probablemente del inconsciente colectivo, despertando un sentimiento de incertidumbre y una alerta mundial.
El virus hizo una rápida zoonosis (Zhang et al., 2020a), se volvió altamente contagioso dentro de la población humana (Chan et al, 2020a) y, desde que se reportó el 2019-nCoV en China, ha venido evolucionado y se ha extendido rápidamente por todo el mundo convirtiéndose en una amenaza global. En tal sentido, el 11 de marzo de 2020 la OMS decidió declarar oficialmente el COVID-19 como una pandemia, la quinta de los últimos cien años, después de la gripe española de 1918 (Belser & Tumpey, 2018; Johnson et al., 2002) causada por el virus H1N1, que se estima produjo entre 20 y 50 millones de muertes; la gripe asiática de 1957 por H2N2 (Viboud et al., 2016), con 1,5 millones de fallecimientos; la gripe de Hong Kong de 1968 por H3N2 (Honigsbaum, 2020), con por lo menos 1 millón de decesos; y la gripe pandémica de 2009 por H1N1 (Jhaveri, 2020), que produjo 300,000 muertes. Ahora bien, la historia de las grandes pandemias que han afectado a la humanidad se extiende mucho más allá de los últimos cien años, empero, como se ha señalado, las últimas cinco son las que se han registrado sistemáticamente siguiendo las herramientas del método científico.
Este artículo responde a la necesidad de presentar a un público amplio los aspectos tecnocientíficos más relevantes del COVID-19, y para ello se ha expuesto de forma concisa pero sistemática una serie de elementos de información sobre el nuevo coronavirus y sus implicaciones en la salud humana. Esto no es tarea fácil, pues la alerta social que se ha producido como consecuencia de la pandemia tiene a la comunidad científica internacional haciendo un gigantesco esfuerzo por desarrollar estudios sobre la patogenicidad, diseño de antivirales y el desarrollo de vacunas contra el 2019-nCoV. En consecuencia, el avance en el estudio de la microbiología del SARS-CoV-2 ha crecido exponencialmente y, mientras se escribe el presente artículo se están produciendo nuevos hallazgos. Sin embargo, se juzga necesario que la comunidad académica y profesional que no es especialista en áreas relacionadas con la biomedicina, tenga suficiente información para crearse un criterio a fin de visualizar que el COVID-19 se quedará perviviendo con la humanidad y es necesario aprender a enfrentar esta microscópica pero poderosa fuerza de la Naturaleza.
Metodología
En este artículo desarrolló una investigación cualitativa-documental sobre el COVID-19. Se utilizaron bases de datos reconocidas tales como: Web of Science (ISI web), ScienceDirects®, SciFinders®, PubMeds®, y Google Scholar™, haciendo énfasis en áreas relacionadas con química, bioquímica o biomédica, y utilizando como entrada o palabras clave: Coronavirus, COVID-19, SARS-CoV-2, 2019-nCoV, viral pneumonia, pandemic, y otros términos análogos. Se empleó como rango el período comprendido entre el 01/01/2020 al 15/10/2020, tomando como referencia inicial los reportes originales del virus de Wuhan.
Resultados y Discusión
Pandemias, plagas y pestes a lo largo de la historia
La humanidad, a través de su historia, ha convivido con diversos tipos de epidemias o pandemias a las que entró a dar el apelativo de pestes o plagas según el grado de devastación que provocaban (Kiple, 1993). Desde un punto de vista epidemiológico, se deben entrar a considerar tres categorías: (1) El brote, que se caracteriza por la aparición repentina en un lugar y momento específico de una enfermedad debida a una infección. En este caso se puede tomar como ejemplo una intoxicación alimentaria que aparece en una comunidad y tiene una duración de dos a tres días. También entran en esta categoría los brotes de meningitis o sarampión que pueden tener una duración de dos a tres meses. (2) La epidemia, que corresponde a una enfermedad que se propaga de manera progresiva debido a la aparición de un brote fuera de control, y se mantiene en el tiempo. En una epidemia se observa el aumento progresivo en el número de casos dentro de un área geográfica específica y mucho más extensa. Actualmente existen modelos que permiten entrar a describir la progresividad matemática de una epidemia sobre la base del clásico modelo SIR y las modificaciones MSEIR y SEIR entre otros (Bichara et al., 2015; Hethcote, 2000; Arino et al., 2007; Brauer, 2017). (3) La pandemia, que llega a ser declarada cuando se cumplan dos criterios: (a) Que el brote epidémico aparezca en más de un continente y, (b) Que los casos registrados en una nación específica no tengan una fuente foránea, es decir, que no sean importados, sino que la trasmisión sea de carácter comunitaria (Morens et al., 2009). En el caso d -19, durante las primeras semanas, y mientras que los casos que aparecían principalmente en Europa eran importados y el foco epidémico se encontraba ubicado en Wuhan, China, la situación tenía el estatus de epidemia, pero en la misma medida que el virus 2019-nCoV se extendía y comenzaron a identificarse casos comunitarios en varios continentes, la OMS se vio en la obligación de declarar una situación de pandemia.
Haciendo un poco de historia, se puede mencionar dentro de los primeros episodios epidémicos con registro histórico a la “plaga de Atenas” (Soupios, 2004), que apareció en esta ciudad alrededor del año 430 antes de J.C. Esta plaga, mencionada por Hipócrates (460-377 antes de J.C.) y Tucídides (460-395 antes de J.C.), se desarrolló mientras la ciudad estaba en estado de sitio por parte del ejército espartano, algo que favoreció su rápida propagación. Se estima que el primer brote comenzó en Etiopía y desde allí se extendió por Egipto, cruzando el mediterráneo hasta llegar a Atenas. Tucídides describió una enfermedad que se caracterizaba por la repentina aparición de fiebre alta, sed intensa (polidipsia), lengua y garganta sangrante, piel enrojecida y amoratada que daba lugar a la aparición de lesiones ulcerosas (Parry, 1969). Por las características clínicas de la enfermedad, se ha especulado sobre la posibilidad de que se trataba de una forma de sarampión o viruela (Cunha, 2004).
El imperio romano también vio aparecer y desaparecer varias epidemias y, a pesar de que las condiciones sanitarias en Roma eran avanzadas para su época, debido a sus acueductos y alcantarillados, desde el siglo III después de J.C., y al mismo tiempo que se producía la decadencia del sistema político y social, surgían brotes de diversas enfermedades (Brothwell & Sandison, 1967).
Los problemas epidemiológicos se agravaron con la llegada de las invasiones bárbaras, pues los pueblos de Asia Central comenzaron a moverse hacia el oeste, desencadenando un fenómeno de migraciones que afectaron a las poblaciones euroasiáticas. Estos pueblos llevaron a Europa enfermedades hasta ese momento desconocidas para el sistema inmunológico de los romanos. Se puede decir que las “calzadas” construidas por los romanos a lo largo y ancho del imperio, sirvieron de camino a virus y bacterias que utilizaron como vehículo a las caravanas comerciales, a los pueblos migrantes, y a las legiones de soldados que debían desplazarse cumpliendo funciones de seguridad y defensa de las fronteras romanas. En este escenario apareció la “peste antonina”, llamada así por el nombre del emperador Marco Aurelio Antonino (Littman & Littman, 1973).
La enfermedad surgió en el año 164 entre las tropas romanas que hicieron campaña en lo que hoy es Irak y se extendió hasta el año 189. La pandemia devastó toda la extensión del imperio romano, desde sus fronteras orientales en Arabia hasta sus fronteras occidentales en el río Rin y la Galia (Alemania y Francia), con una alta tasa de mortalidad que afectó a toda la población, desde los nobles hasta los plebeyos y esclavos (Fears, 2004). El famoso médico romano Galeno (129-199) describió las características de la misma y mencionó que los síntomas iniciales eran fiebre alta, inflamación de boca y garganta, polidipsia o sed intensa, diarrea y, alrededor del noveno día una erupción cutánea, el último signo antes de producirse el fallecimiento del enfermo. La descripción de Galeno lleva a pensar que podía tratarse de una especie de viruela que tuvo como origen Asia central y el norte de China.
Entre los años 249 y 270 se desarrolló otro episodio pandémico de gran impacto, que fue descrito por el obispo Cipriano de Cartago (200-258) (Harper, 2015). La enfermedad comenzaba con una fiebre muy alta y diarrea intensa acompañada de vómitos, polidipsia, garganta ulcerada y la aparición de una gangrena en manos y pies. De manera similar a lo observado en el caso ateniense, el primer brote comenzó en el Cuerno de África, muy probablemente en Etiopía, una región donde un virus pudo pasar con facilidad a los humanos haciendo una zoonosis, y desde allí se desplazó por Egipto y las colonias romanas del norte de África, avanzando con gran rapidez, hasta adquirir la categoría de pandemia, y su contagio ocurría no solo de persona a persona sino a través del contacto con las prendas u objetos de la persona enferma. Según los registros históricos, la patogenicidad fue de una magnitud tal que el número de muertos era mayor que los sobrevivientes responsables darles sepultura (Cartwright, 1993). De acuerdo al obispo Cipriano la primera fase u oleada de esta pandemia duró dieciséis años, tiempo en el cual la humanidad se vio obligada a cambiar su forma de vida. Con altos y bajos, esta enfermedad parece haber persistido en Europa durante los siguientes tres siglos, y sus consecuencias económicas y sociales seguramente fueron uno de los factores que contribuyeron a la caída del sistema imperial romano.
En el año 542 surgió la que se conocerá como tercera gran plaga de la antigüedad clásica, la “plaga de Justiniano” (Allen, 1979) que, por su magnitud será considerada una de las pandemias más letales que conoció la humanidad luego de la plaga de Atenas y la peste antonina. Según el cronista Procopius (500-560) el primer brote ocurrió en la ciudad de Pelusium, en el Bajo Egipto, y se extendió por Palestina y el Medio Oriente y, para el año 558 cuando aparecieron los primeros contagios en Bizancio, ya tenía carácter pandémico. Una fiebre alta y durante los primeros días de una severa inflamación de los ganglios linfáticos (lo que se conocerá como bubones), serán los síntomas de la gran “plaga” propiamente dicha, que corresponde a la “peste bubónica” causada por la bacteria Yersinia pestis. Esta enfermedad, que provoca la muerte al quinto día de manifestar la sintomatología, fue recurrente hasta el año 590.
La plaga por Y. pestis fue una de las enfermedades más importantes, pues muy pocos microorganismos han matado hasta un tercio de la población mundial durante una pandemia, cambiado el curso de la historia (Raoult et al., 2013). Esta enfermedad reapareció en el siglo XIV adquiriendo la denominación de “peste negra”, y fue bien descrita por el cirujano francés Guy de Chauliac (1298-1368), médico de la corte de los papas en Aviñón, quien la asoció a una adenitis (bubón) con altísima tasa de mortalidad.
El nuevo coronavirus del COVID-2019 y sus características
El SARS-CoV-2 no es más que una envoltura (saco o bolsa) esférica y microscópica de aproximadamente 120 nm de diámetro que contiene una única molécula de ARN en la cual se encuentra codificado todo su genoma (figura 1). El nuevo visitante que irrumpió en la antroposfera pertenece a la subfamilia Coronavirinae, familia Coronaviridae, orden Nidovirales (Perlman & Netland, 2009), y se caracteriza por unas proteínas que brotan como espigas desde su superficie (proteína S), se alargan y terminan en una en una punta trimérica; estas proteínas de manera similar a las astas de una corona, permiten al 2019-nCoV impulsarse e interaccionar, como si fuera una “maquina molecular”, con la célula de quien se convertirá en su hospedero.
El análisis bioinformático del genoma de un virus encontrado en un paciente COVID-19 (Chan et al., 2020b) permitió comparar genéticamente al 2019-nCoV frente a otros genomas de coronavirus. En total el genoma del nuevo coronavirus posee 29.903 nucleótidos (Wu et al., 2020a), de los cuales el 89% tienen similitud con el encontrado en el coronavirus del murciélago (SARS-like-CoVZXC21), asimismo tiene un 82% de semejanza con el coronavirus humado previamente reportado, el SARS-CoV que también surgió en China entre 2002-2003 y causó una epidemia por síndrome respiratorio agudo severo que afectó a unas 8.000 personas, con una tasa de letalidad del 10% (Luk et al., 2019). El estudio detallado del genoma realizado sobre aquellas zonas que almacenan la información de las estructuras más importantes del virus: La proteína S (spike protein), la envoltura, la membrana glicoproteica, la nucleoproteína o nucleocápside, y el gen orf1a/b, reiteran la cercanía del 2019-nCoV con diversos coronavirus encontrados en murciélagos, civetas, camellos y finalmente humanos. En general, los coronavirus, que se identificaron por primera vez hace setenta años, pero que no recibieron notoriedad hasta la epidemia del 2002-2003, ocasionan principalmente infecciones respiratorias y del tracto gastrointestinal, y están genéticamente clasificados en cuatro géneros (Li, 2016): Alfacoronavirus (α-CoV), Betacoronavirus (β-CoV), Gammacoronavirus (γ-CoV) y Deltacoronavirus (δ-CoV). Por sus características genéticas el 2019-nCoV es un betacoronavirus.
Es interesante mencionar que la proteína S del nuevo coronavirus 2019-nCoV-2 (humano), es más larga que sus homologas del SARS-CoV (murciélago) y MERS-CoV (camello). El 2019-nCoV penetra en la célula empleando como receptor a la enzima convertidora de angiotensina 2 (ACE-2) y, a pesar de que la estructura de la glicoproteína de la envoltura es ligeramente diferente, se ha demostrado in vitro que el ACE-2 sigue siendo un receptor válido para el SARS-CoV-2 (Zhou et al., 2020a).
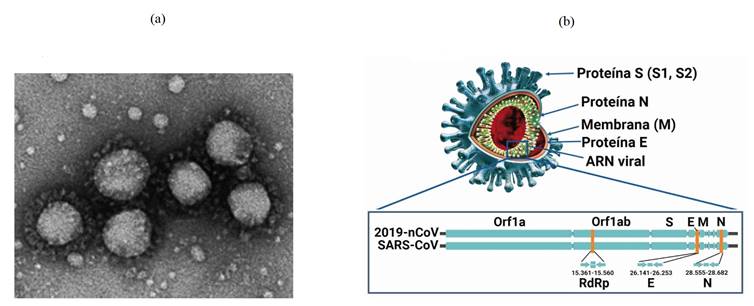
Figura 1: (a) Imagen por microscopía electrónica de barrido del 2019-nCoV asilado de un paciente de la primera oleada de contagios de SARS-CoV-2 en Wuhan, Hubei, China (Jiang, 2020). (b) Representación esquemática de las partes más importantes del nuevo coronavirus: La proteína E (envoltura vírica), la proteína S (la espiga de la corona), la proteína N (nucleoproteína), la membrana glicoproteica (envoltura del virus) y el ARN viral. Adicionalmente, se pueden apreciar en la parte inferior una ampliación de las posiciones relativas de las proteínas S, N y E, de la membrana (M) en el genoma del nuevo coronavirus 2019-nCoV y del SARS-CoV (figura adaptada de Kubina & Dziedzic, 2020 y Corman et al., 2020).
Mecanismo de trasmisión y poblaciones susceptibles del COVID-19
El COVID-19 se puede propagar a través de microgotas de origen respiratorio durante el contacto cercano, en virtud de que el virus tiene un predominio en el tracto respiratorio superior. En consecuencia, es posible adquirir COVID-19 cuando una persona sana se encuentra cerca de una persona infectada que tose, estornuda o incluso habla, luego de una exposición inicial sin tomar precauciones, del orden de 15 minutos (este tiempo puede ser continuo o intermitente), a menos de 2 metros de distancia (6 ft), condición que define el “contacto estrecho” según el Centro para el Control y la Prevención de Enfermedades de Estados Unidos (CDC). Luego de este primer contacto pueden pasar hasta 14 días antes de que una persona desarrolle los síntomas, sin embargo, existen reportes que indican que la mediana del tiempo desde que ocurre la exposición hasta el inicio de los síntomas puede ser del orden de cuatro a cinco días (CDC, 2020). Quizá uno de los mayores riesgos que se observa en esta pandemia lo constituye el hecho de que más del 80% de las personas infectadas son asintomáticas o tienen síntomas leves (Wu et al., 2020b), y por lo tanto son capaces de propagar el virus sin saberlo, aunque el riesgo de transmisión es mayor en los pacientes que manifiestan los síntomas de la enfermedad. En virtud de que la mayoría de los casos leves o asintomáticos no se informan, es difícil identificar y establecer medidas para contener las áreas de alto riesgo. Como es bien conocido, las pautas de manejo han recomendado lavarse las manos con frecuencia, evitar el contacto estrecho y hacer uso racional de una mascarilla adecuada (Tso & Cowling, 2020; Feng et al., 2020). En tal sentido, en primer lugar, se tiene la mascarilla N95 que ofrece la posibilidad de filtrar hasta un 95% de las microgotas (<5 μm), es decir, aerosoles; sobre las mascarillas quirúrgicas existe una recomendación similar. Por otro lado, se presentan las mascarillas KN95, que se estiman filtra entre un 53% y 85% (Dugdale & Walensky, 2020). No obstante, la medida más efectiva siempre será quedarse en casa y mantener el distanciamiento físico.
A pesar de todos los mecanismos de contención, en una situación pandémica y frente a un virus como el 2019-nCoV, el riesgo siempre es alto y, aunque todos los seres humanos son capaces de contraer el COVID-19, existen poblaciones que tienen un mayor riesgo de desarrollar la enfermedad con síntomas más graves debido a condiciones de salud subyacentes. Se estima que 1.700 millones de personas, que comprenden el 22% de la población mundial, tienen al menos una afección subyacente que aumenta la probabilidad de tener síntomas graves por COVID-19 (desde <5% de los menores de 20 años hasta >66% de los mayores de 70 años), así mismo, aproximadamente 350 millones de personas (4% de la población mundial) tienen un alto riesgo de llegar a un cuadro grave de COVID-19 que va requerir el ingreso a un centro hospitalario (que van desde <1% de los menores de 20 años a aproximadamente el 20% de las personas mayores de 70 años) (Clark et al., 2020). En el caso de los adultos mayores (13,4% de los pacientes mayores de 80 años) el riesgo es mayor debido a un sistema inmunológico debilitado y a las patologías preexistentes que aceleran la infección viral (Verity et al., 2020; Rothan & Byrareddy, 2020). El ejemplo emblemático lo encontramos en Europa y el caso español e italiano donde se observó un alto número de brotes de COVID-19 en centros de atención al adulto mayor con altas tasas de morbilidad y letalidad. Las personas de cualquier edad pero que han desarrollado afecciones médicas subyacentes tales como enfermedades cardiovasculares, hipertensión, diabetes, enfermedades respiratorias crónicas, enfermedad renal crónica y cáncer, también tienen un mayor riesgo de sufrir desenlaces fatales.
Los pacientes inmunodeprimidos, ya sea por condiciones preexistentes o tratamientos médicos como en el caso de los que están sometidos a quimioterapia (Xia et al., 2020) tienen una capacidad reducida para resolver infecciones virales, lo que hace que estos pacientes sean más vulnerables al 2019-nCoV (Raifman & Raifman, 2020) y tengan peor prognosis (Miyashita et al., 2020). En el caso de los pacientes que son portadores del VIH, con la enfermedad bien controlada, no tienen riesgo de COVID‐19 mayor que la población general, pero lo que no está claro es si aquellos pacientes mal controlados tienen peores resultados (Cooper et al., 2020). Otros estudios introducen nuevos criterios de riesgo como niveles de colesterol elevado, tabaquismo, un anormal índice de masa corporal, inadecuados niveles de vitamina D, mala alimentación, estatus socioeconómico e incluso origen étnico (Raisi-Estabragh et al., 2020).
Es importante señalar que el COVID-19 se puede expresar con sus síntomas más graves en personas sin factores de riesgo conocidos y, no obstante, la existencia de los grupos o poblaciones de riesgo no existe edad o grupo etario que pueda considerarse inmune a la enfermedad y, en consecuencia, todos sin distinción deben tomar las medidas de protección.
El COVID-19, síntomas y patogenicidad
La “patogenicidad” de un virus define la gravedad de los problemas de salud que se producen cuando una persona se convierte en el hospedero, especialmente cuando se compara entre diferentes virus; en tal sentido, el virus del ébola (EVE) es más patógeno que el sarampión. Por otro lado, la “virulencia” hace referencia a la gravedad de la enfermedad causada por diferentes variaciones de un mismo microorganismo o, en otras palabras, la cantidad (carga viral) de virus que es necesaria para producir un problema grave de salud o incluso la muerte (Collier & Oxford, 2016). Por ejemplo, dos cepas diferentes del virus del herpes simple (HSV) inoculadas en la piel de un ratón pueden causar lesiones cutáneas, sin embargo, 10 viriones o partículas de la cepa A de este virus puede matar al ratón, mientras que para que produzca el efecto fatal se necesitan 10.000 viriones de la cepa B; en consecuencia, la cepa A es mil veces más virulenta que la cepa B.
Ahora bien, lo que debe quedar claro es que, una vez que ocurre el contagio, se desencadenan una serie de eventos bioquímicos, es decir, un conjunto de interacciones entre el virus y las células dentro de las cuales va a ocurrir su replicación, y será esto último lo que tendrá una importancia decisiva a la hora de determinar si va a desarrollar una infección, de que tipo será esta, y cuál será el resultado final para la salud del hospedero. Recordemos que un virus buscará un hospedero con el objetivo de reproducirse, puesto que el camino evolutivo seguido por éstos, les condujo a una extraordinaria pero eficiente simplicidad y los llevó a prescindir de todo los sistemas microbiológicos necesarios para replicarse de manera independiente, algo que si poseen las células (incluyendo bacterias), las cuales pueden replicarse de manera eficiente. Dicho de otra manera, los virus van a parasitar a una célula (Carter & Saunders, 2007) y, puesto que su objetivo es mantenerse en el tiempo, deben valerse de las células de su hospedero.
En el caso de los coronavirus, se ha observado que se replican primero en las células epiteliales de los tractos respiratorio o entérico (envolturas del esófago, el estómago, el intestino delgado y el colon). Debido a que los coronavirus están envueltos (ver figura 1), los viriones son menos estables en el medioambiente que la mayoría de los virus no envueltos, de allí que sean susceptibles a sustancias tensoactivas como el jabón (surfactante) que desestabilizan su membrana. Aunque la transmisión se encuentra asociada a un contacto cercano entre hospederos, el SARS-CoV-2 es sorprendentemente estable en superficies ambientales (Burrell et al., 2017) razón por la cual higienizar es tan importante. La patogenicidad y la respuesta inmune a los coronavirus se ha estudiado más en las infecciones por coronavirus en animales y, en este orden de ideas, el virus de la hepatitis del ratón (MHV), que es un betacoronavirus similar al 2019-nCoV, abarca un conjunto de cepas que causan infecciones del tracto gastrointestinal, hepáticas, respiratorias o del sistema nervioso central. Debido a que la enfermedad neurológica causada por el MHV simula la esclerosis múltiple en humanos, se han realizado estudios detallados al respecto (Coleman & Frieman, 2014).
Para causar la enfermedad, el coronavirus, y en general cualquier virus, tiene que superar una serie de obstáculos, que varían según sus características y según su hospedero. En este orden de ideas, los eventos que suceden son: (1) Invadir el hospedero; (2) Establecer una cabeza de puente mediante la replicación en células susceptibles en el sitio de inoculación o de contacto; (3) Superar las defensas locales, por ejemplo, linfocitos, macrófagos e interferón (IFN); (4) Propagarse desde el lugar de la inoculación a otras áreas, normalmente a través del torrente sanguíneo; (5) Incrementar su replicación en su área objetivo, ya sea localizada (como en el caso de la conjuntivitis por adenovirus) o generalizada (como ocurre con el sarampión) y, finalmente, (6) Salir del hospedero en cantidades lo suficientemente grandes como para infectar a otros hospederos susceptibles y así asegurar la pervivencia. Como se puede apreciar, el virus tiene una especie de “plan de ataque” que le permite invadir a un hospedero en particular y luego propagarse indefinidamente hasta que se den las condiciones para que sea contenido y, como puede inferirse, algunas de estas etapas dependen de las características del virus, de la forma en que ocurre la interacción virus/hospedero, tanto dentro de las células individuales como del organismo en general, y toma en cuenta las defensas inmunitarias o la resistencia que le pueda hacer el hospedero.
A partir de la descripción anterior, es importante conocer las vías que el virus utilizará para realizar su plan de ataque, en este caso la piel, las membranas y mucosas, la ruta respiratoria, pero también la gastrointestinal o la vía genital, solo por mencionar algunas.
En consecuencia, el virus puede ingresar a través de una superficie epitelial, donde experimentará una replicación limitada, a partir de allí los viriones migran a los ganglios linfáticos donde algunos son atrapados por los macrófagos que, en una primera instancia, consiguen detenerlos, pero otros muchos ingresan al torrente sanguíneo. Esta etapa constituye una viremia primaria, que a veces da lugar a malestar general y fiebre. Una vez en el torrente sanguíneo, el virus gana acceso a los órganos reticuloendoteliales grandes (bazo, hígado y médula ósea), donde se amplifica la capacidad de replicarse, y por lo tanto se produce una gran cantidad de viriones que nuevamente, pero de forma masiva, van haciendo uso del torrente sanguíneo para diseminarse, causando la viremia secundaria. Desde el torrente sanguíneo, el virus alcanza su meta, algún órgano específico del cuerpo cuya identidad dependerá del propio virus (tropismo tisular). Esto último va a determinar las características clínicas de la enfermedad. Este conjunto de etapas en las que el virus va invadiendo a su hospedero, justifican que las viremias tomen un tiempo hasta que se manifiesta la enfermedad, lo que explica la necesidad de un período de incubación del orden de 2 semanas.
En el caso específico del 2019-nCoV, se han reportado con detalle los síntomas que se producen con motivo de su contagio, entre los cuales se destacan fiebre, cefalea, dolor de garganta, anosmia (pérdida del sentido del olfato), astenia (debilidad general), síntomas digestivos como dolor abdominal, diarrea, náuseas, estreñimiento, y anorexia, síntomas hepáticos, en este caso la hipertransaminasemia, y finalmente síntomas propiamente respiratorios comenzando por la tos, que puede llegar a una neumonía atípica y severa, que es la principal causa de muerte en los pacientes con COVID-19. Esta neumonía atípica implica una fuerte dificultad respiratoria debido a la inflamación en el revestimiento de los pulmones causada por la “tormenta de citoquinas”, que se observa en la insuficiencia respiratoria y en todos los casos de no supervivencia. La inflamación resultante en los pulmones viene a dar acceso a un numeroso conjunto de infecciones asociadas con insuficiencia de órganos diana de la coagulopatía. De hecho, el COVID-19 coloca de manifiesto infecciones desatendidas o subyacentes que nunca se expresaron con anterioridad, pero que ahora surgen con fuerza en los pacientes, por ejemplo, alteraciones cutáneas tipo psoriasis (Pigliacelli et al., 2020). En tales condiciones sobreviene un choque séptico que genera una falla multiorgánica y finalmente la muerte.
El diagnóstico del COVID-19. La RT-PCR y las pruebas rápidas
Como se ha señalado, el orden en el cual los síntomas del coronavirus aparecen inicialmente sería fiebre, cefalea, dolor de garganta, anosmia, astenia, dolor muscular y luego náuseas y/o vómitos y diarrea. Conocer el orden de aparición de los síntomas es importante especialmente para que los afectados por el COVID-19 puedan tomar la decisión de practicar un autoaislamiento o buscar atención médica rápidamente, lo cual es importante debido a que se ha observado que los pacientes pueden llegar a desarrollar el síndrome respiratorio agudo dentro de los dos días posteriores al ingreso hospitalario, lo que requiere soporte ventilatorio, una fase donde la mortalidad tiende a ser alta (Huang et al, 2020). Identificar el orden de aparición de las características clínicas ayuda también a liberar un poco de presión sobre los centros de salud que obviamente no estaban preparados para atender una pandemia de esta naturaleza y los problemas éticos que implica la clasificación (triage) de los pacientes pues sólo habrá un número limitado de camas, ventiladores, enfermeras y médicos disponibles (White & Lo, 2020; Tabery & Mackett, 2008). Por otro lado, conocer en detalle toda la sintomatología colabora con los profesionales de la salud que pueden descartar otras enfermedades y establecer un plan de acción sobre el tratamiento de cada paciente (Larsen et al., 2020) e intervenir de manera temprana, antes de que aparezca el síndrome respiratorio agudo. El orden de aparición de los síntomas es especialmente importante en momentos en los cuales se observan ciclos superpuestos de enfermedades como la gripe que coinciden con el COVID-19, puesto que la fiebre y la tos se asocian con una variedad de enfermedades respiratorias.
Uno de los principales métodos de diagnóstico del SARS-CoV-2 se basa en la reacción en cadena de la polimerasa con transcripción inversa en tiempo real, RT-PCR (real-time reverse-transcriptase-polymerase chain reaction) y en los estudios por imagenología (radiografía y tomografía computarizada) (Udugama et al., 2020; Long et al., 2020; Xiao et al., 2020; Corman et al., 2020). Adicionalmente, se encuentran desarrolladas las pruebas de inmunodiagnóstico, basadas en la presencia de anticuerpos, las denominadas pruebas rápidas, o kits de pruebas de diagnóstico, POC (point-of-care), que están desempeñando un papel importante. En principio, estas pruebas rápidas fueron recomendadas por la OMS solo para fines de investigación, sin embargo, durante el primer semestre de 2020 muchos países adoptaron la decisión de implementar de manera preventiva el uso de estas pruebas rápidas con la esperanza de aumentar el número y la velocidad de las pruebas (Kubina & Dziedzic, 2020). No obstante, muy pronto se comenzaron a observar variaciones entre los resultados, generando un debate sobre la sensibilidad y especificidad de las pruebas de inmunodiagnóstico (Keni et al., 2020). En las pruebas inmunológicas, es mucho más adecuado analizar los anticuerpos producidos como respuesta al virus, en lugar de analizar la presencia de proteínas virales, especialmente si se considera que los anticuerpos pueden estar presentes en mayor cantidad y durante un período más largo. No obstante, una prueba rápida nunca podrá sustituir a la prueba a la RT-PCR o a los estudios por imagenología médica.
Existe una tercera clase prueba de diagnóstico para el SARS-CoV-2, una prueba serológica, la tradicional prueba de sangre. Se trata de ensayos basados en la inmunoglobulina (IgG, IgM o ambos), por medio de inmunoabsorbente ligado a enzimas (ELISA), inmunoensayos de flujo lateral o inmunocromatográficos (LFIA), y los inmunoensayos por tecnología quimioluminiscente (CLIA). Estas pruebas son una herramienta útil en el diagnóstico, pero su sensibilidad y la especificidad, las plantea como una alternativa complementaria a la prueba RT-PCR (Bastos et al., 2020).
Tratamientos para el COVID-19. Remdesivir o Veklury®
El SARS-CoV-2 no dispone aún de un único tratamiento, se trata más bien de una estrategia o combinación de tratamientos (Pushpakom et al., 2018) que se aplica según aparecen los síntomas. En tal sentido, la Organización Mundial de la Salud y sus aliados institucionales, desde el mes de marzo de 2020, han patrocinado el “ensayo Solidarity” (WHO, 2020c), un ensayo clínico internacional dirigido a encontrar un tratamiento eficaz para el COVID-19, y constituye uno de los ensayos aleatorizados internacionales más grandes que involucra a 9.000 pacientes en 500 centros hospitalarios de más de 30 países (WHO, 2020d).
El ensayo Solidarity está suministrando importante evidencia sobre la actividad de los antivirales, los antimaláricos y el interferón, entre otros fármacos, en un estudio que está diseñado con la finalidad de reducir en más de un 80% el tiempo utilizado en la “prueba controlada aleatorizada” (RTA por sus siglas en inglés). Hasta el momento, existen más de 1.200 estudios registrados en la base de datos de ensayos clínicos administrada por la Biblioteca Nacional de Medicina y los Institutos Nacionales de Salud de EE.UU. (clinicaltrials.gov). Naturalmente, los estudios se concentran en medicamentos antivirales, pero también en medicamentos antipalúdicos y diferentes formas de oxigenoterapia. La mayoría de los ensayos tratan de analizar la incidencia del medicamento en el estado clínico del paciente, en lo que respecta a la reducción de la carga viral, el tiempo de recuperación y la reducción de las tasas de mortalidad, cubriendo los casos graves y leves. Además del ensayo Solidarity existe el “ensayo Recovery”, que involucra más de 12.000 pacientes en 176 centros hospitalarios, lo cual significa que entre los ensayos Solidarity y Recovery se agrupa una población de 20.000 pacientes en casi un millar de instituciones médicas.
A pesar del inmenso esfuerzo científico y tecnológico, hasta el momento de los aproximadamente 2.000 medicamentos probados en estudios contra el COVID-19 (covid-trials.org), la mayoría no han dado resultados positivos (Tikkinen et al, 2020), excepto en el caso del antiviral Veklury® (remdesivir, código de desarrollo GS-5734), que comenzó a arrojar algunos resultados preliminares de actividad (Grein et al., 2020). En el “ensayo ACTT-1” llevado a cabo por el Instituto Nacional de Alergias y Enfermedades Infecciosas de EE.UU. (NIAID), que involucró a más de 1.000 pacientes, donde Veklury® (figura 2) demostró un promedio de tiempo de recuperación del COVID-19 de 10 días y la mortalidad general a los 29 días fue del 11% (FDA, 2020a). Si bien es cierto que en el ensayo Solidarity no se observaron resultados tan alentadores, tampoco llegaron a refutar los hallazgos del ensayo ACTT-1 en cuanto al beneficio para los pacientes, razón por la cual el Veklury® - remdesivir (NDC 61958-2902-2) fue finalmente aprobado por la Administración de Alimentos y Medicamentos de los EE.UU. (FDA por sus siglas en inglés) como el primer antiviral para el tratamiento del COVID-19 (FDA, 2020b), un medicamento fabricado por Gilead Sciences, empresa estadounidense de biotecnología, que ha elaborado otros medicamentos antivirales para el ébola, el VIH, la hepatitis B o la influenza.
Tratamientos para el COVID-19. Cloroquina y la dupla lopinavir/ritonavir
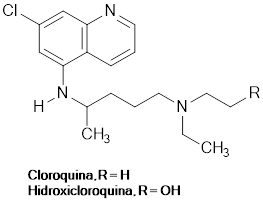
Los ensayos realizados hasta el momento, entre ellos el Solidarity, no han arrojado evidencia suficientemente en favor de un tratamiento contra el COVID-19 basado en antimaláricos como la cloroquina (CQ) y la (HCQ) hidroxicloroquina (figura 3), El uso de la CQ y la HCQ se remonta a mediados del siglo XX (Meshnick & Dobson, 2001), pues se trata de dos fármacos indicados en el tratamiento tanto de la malaria como de algunas enfermedades virales, autoinmunes (Savarino et al., 2003) y la artritis reumatoide (Schrezenmeier et al., 2020). La CQ y la HCQ tienen mecanismos de acción similares, pues su actividad farmacológica incluye propiedades lisosomotrópicas, previenen la conversión del grupo hemo tóxico en hemozoína no tóxica, logrando, por tanto, niveles excesivos de toxicidad dentro del parásito Plasmodium; adicionalmente, ambos medicamentos tienen efectos inmunorreguladores y regulan negativamente las citoquinas proinflamatorias.
CQ y HCQ mostraron inicialmente efectos antivirales contra SARS-CoV-2 in vitro (Liu et al., 2020a), inhibiendo la replicación del virus (Wang et al., 2020), y, por otro lado, se reportó que la administración de antibióticos como azitromicina junto con hidroxicloroquina podría ayudar en el tratamiento de superinfecciones bacterianas observadas en los pacientes hospitalizados (Gautret et al.2020).
No obstante, la evidencia acumulada hasta el momento exige que se sigan realizando estudios, pues lo que se observa es que el uso de CQ y HCQ en COVID-19 ha sido objeto de una controversia a nivel mediático (Cross, 2020) y, por un lado, estos fármacos son promocionados como una especie de panacea, mientras que en el otro extremo son catalogados como inútiles y hasta peligrosos (Khuroo et al., 2020). En tal sentido, no está clara la dosificación óptima, por ejemplo, en el caso de CQ se indica 500 mg por vía oral una o dos veces al día durante 5 a 10 días (Colson et al., 2020) y, para HCQ, una dosis de 400 mg dos veces al día el día, luego 200 mg dos veces al día en los siguientes 2-5 días (Yao et al., 2020).
Es importante señalar que, el 28 de marzo de 2020, la FDA autorizó el uso CQ y HCQ en las emergencias para pacientes hospitalizados con COVID-19 (Magagnoli et al., 2020), sin embargo, revocó dicha autorización debido a informes de problemas del ritmo cardíaco.
En todo caso, parece no existir evidencia de que el uso de HCQ redujera el riesgo de ventilación mecánica en los pacientes y, por otra parte, los efectos colaterales (oculares y cardiovasculares) de un tratamiento con CQ y HCQ deben ser vistos con cautela (Chatre et al., 2018; Stokkermans et al., 2019; Marmor et al., 2002).
Algo que está claro es la necesidad de investigación al respecto, pues la familia de fármacos antipalúdicos es extensa y, además de CQ y HCQ se deben estudiar los modernos antimaláricos (Ashton et al., 2019), incluyendo compuestos complejos de metales de transición (Navarro et al., 2010) o metaloantimaláricos (Salas et al., 2013) y compuestos organometálicos (Contreras et al., 2018; Contreras et al., 2012; Aranguren & Contreras, 2010).
El medicamento antiviral Kaletra® (Corbett et al., 2002) es una combinación de los fármacos lopinavir (Stone et al., 2000) y ritonavir (Kempf et al., 1998), que se administra por vía oral y se encuentre aprobado por FDA como tratamiento de infecciones por VIH. El lopinavir inhibe las proteasas del virus de la inmunodeficiencia humana tipo 1 y 2 (VIH-1 y VIH-2), dando como resultado un virus inmaduro no infeccioso (Croxtall & Perry, 2010). Debido a su baja biodisponibilidad oral y su alto porcentaje de biotransformación, el lopinavir se coadministra con ritonavir para prolongar los niveles dentro del organismo, luego, el ritonavir actúa como un refuerzo farmacocinético del lopinavir (Cvetkovic & Goa, 2003).
En resumen, el efecto antiviral del lopinavir responde a su capacidad de prevenir la infección de células susceptibles. La asociación lopinavir/ritonavir ha sido probado antes de la pandemia sobre los coronavirus SARS-CoV (Barrila et al., 2006) y MERS-CoV (Rabaan et al., 2017), apuntando una posible actividad inhibidora de la proteasa tipo 3-quimiotripsina, que sería un objetivo clave para evitar la replicación de coronavirus humanos. Ya en el marco de la pandemia del COVID-19, se realizó un ensayo in vitro que sugiere una inhibición de la replicación del SARS-CoV-2 (Choy et al., 2020), e incluso se llegó a sugerir su aplicación en las etapas iniciales de la enfermedad para reducir la mortalidad (Jin et al., 2020). Sin embargo, la evidencia no es suficiente como para indicar que exista un beneficio para los pacientes (Cao et al., 2020a; Vijayvargiya et al., 2020; Li et al., 2020).
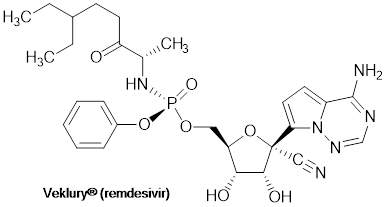
Figura 2: Estructura química del Veklury® (remdesivir, código de desarrollo GS-5734), primer medicamento antiviral aprobado por la FDA como tratamiento para el COVID-19. Se trata de un profármaco monofosforoamidato y nucleosídico análogo de la adenina. Dentro de las células, se convierte en un trifosfato, que es la forma farmacológicamente activa capaz de inhibir la ARN polimerasa viral dependiente de ARN (RdRp) (Eastman et al., 2020).
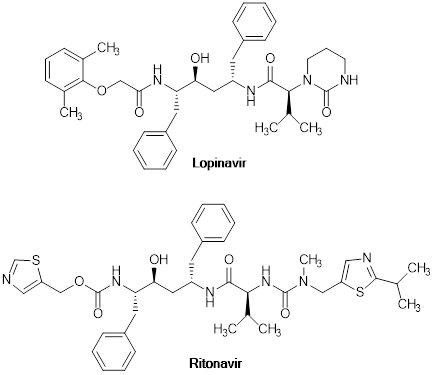
Figura 4: Estructura química de los fármacos lopinavir (ABT-378) (Stone et al., 2000) y ritonavir (ABT-538) (Kempf et al., 1998). La combinación de ambos responde al fármaco Kaletra® Figura 4 (Corbett et al., 2002).
Otros tratamientos farmacológicos propuestos para el COVID-19
Actualmente estamos en presencia de una carrera contra reloj que tiene como protagonistas a científicos de todo el mundo y al nuevo coronavirus del COVID-19, se trata de investigar medicamentos que tienen comprobada actividad farmacológica y por tanto puedan ser candidatos en la lucha contra el 2019-nCoV (De Savi et al., 2020). Esta estrategia es más efectiva que emprender el desarrollo de nuevos fármacos, pues llevaría un tiempo que no está disponible; sin embargo, las herramientas tecnocientíficas con las que se cuenta en el campo de la síntesis química de medicamentos, la química farmacéutica y la química computacional aplicada al diseño de fármacos, permiten visualizar poderosas estrategias en este sentido.
Siguiendo en el campo de los antivirales, se probó Vilona® (ribavirina) (Gebeyehu et al., 1985) (figura 5), un análogo de la guanina que tiene la capacidad de inhibir la ARN polimerasa dependiente de ARN o RdRp, y se usa para el tratamiento de la influenza (Gross & Bryson, 2015) o la hepatitis C (Feld & Hoofnagle 2005). Previamente fue probada en los brotes de SARS-CoV de 2003 (Stockman et al., 2006) y MERS-CoV de 2012 (Mo & Fisher, 2016), pero no se llegó a determinar un beneficio terapéutico significativo. A pesar de ello, se realizaron pruebas frente al SARS-CoV-2, que tampoco fueron concluyentes al respecto (Sanders et al., 2020; Khalili et al., 2020).
Avigan® (favipiravir, T-705) (figura 5), es otro antiviral que actúa vía inhibición de RdRp (Wang et al, 2016), y está probado como tratamiento para la influenza (Shaw 2017), el ébola y el norovirus. Las pruebas realizadas hasta el momento indican que favipiravir tiene un efecto superior que la dupla lopinavir-ritonavir (Cai et al., 2020), pues se observó una reducción en la fiebre y la tos, pero el efecto no fue significativo entre los pacientes críticos afectados por el COVID-19 (Chen et al., 2020b), no obstante, estos resultados no son suficientemente significativos como para dar continuidad a los ensayos clínicos (McKee et al., 2020).
Tamiflu® (oseltamivir) (Magano, 2009) mostrado en la figura 5, es el antiviral que se popularizó con motivo de la pandemia de gripe A H1N1 de 2009-2010, fue desde el comienzo un candidato natural a investigar frente a el COVID-2019. Se trata de un fármaco inhibidor de la neuraminidasa eficaz para el tratamiento de la influenza A y B (Jefferson et al., 2014), a pesar de ello los resultados no indican que sea eficaz como tratamiento del COVID-19 (Awasthi et al., 2020; Sanders et al., 2020; Yousefi et al., 2020; Wu et al., 2020b).
Como se ha reiterado, cuando el COVID-19 infecta el tracto respiratorio (superior e inferior), se produce un síndrome respiratorio leve o muy agudo con la consiguiente liberación de citocinas proinflamatorias, incluida la interleucina IL-1 e IL-6, esto significa que las estrategias antiinflamatorias constituyen una valiosa herramienta frente a la enfermedad. De hecho, los estudios encuentran en los pacientes afectados por el SARS-CoV altos niveles de IL-6 y otras citoquinas proinflamatorias, que son la principal causa de la “tormenta de citosinas” o síndrome de liberación de citoquinas (CRS por sus siglas en ingles), que viene a ser la principal causa de muerte en los pacientes más graves por el COVID-19 (Zhang et al., 2020b). En consecuencia, la supresión de estas citoquinas proinflamatorias resulta en un importante efecto terapéutico (Conti et al., 2020) y esto se puede obtener utilizando agentes antinflamatorios como el Actemra® (tocilizumab).
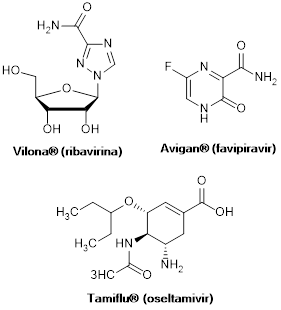
Figura 5: Estructura química de los antivirales: Vilona® (ribavirina) (Gebeyehu et al., 1985); Avigan® (favipiravir, T-705) (Wang et al., 2016) y Tamiflu® (oseltamivir) (Magano, 2009).
Actemra® o RoActemra® (tocilizumab) es un anticuerpo monoclonal humanizado dirigido contra el receptor de la interleucina-6 (IL-6) y está aprobado por la FDA para el tratamiento del síndrome de liberación de citoquinas (SRC) propio de la artritis reumatoide y la artritis idiopática juvenil. Algunos estudios han reportado resultados positivos en pacientes con un cuadro críticos del COVID-19 que tiene niveles altos de IL-6 (Toniati et al., 2020; Farooqi et al., 2020; Khiali et al., 2020; Xu et al., 2020), pero es necesario continuar con los estudios sistemáticos antes de hacer una recomendación y, en tal sentido ya la FDA aprobó un ensayo clínico de fase III (Salvi & Patankar, 2020), pero es necesario tomar en consideración los costos que involucra un tratamiento con este medicamento.
El Kineret® (anakinra), otro fármaco utilizado como tratamiento para artritis reumatoide, que es inhibidor de la interleucina-1, tiene un estatus similar al tocilizumab, con un ensayo en fase II aprobado (Cavalli et al., 2020).
Los interferones de tipo I y tipo III (IFNs) forman parte de un grupo de biomoléculas que poseen propiedades antivirales e inmunomoduladoras (Stark et al., 1998; Samuel, 2001), y dentro de este grupo los interferones tipo I, interferón-α (IFNα), interferón-β (IFNβ), y los tipo III, interferón-λ (IFN- λ), han sido postulados como candidatos en el tratamiento del COVID-19 (Sallard et al., 2020; Mantlo et al., 2020; Portela & Brites, 2020). Los interferones se unen a los receptores de interferón alfa/beta (IFNAR) en la membrana celular, que fosforilan el transductor de señal y activador de la transcripción (STAT1) y otras proteínas de este tipo (STATs). Una vez que la proteína STAT1 alcanza el núcleo celular, activa genes estimulados por interferón (ISG), un proceso cuyo efecto neto es de naturaleza inmunomoduladora y viene a interferir con la replicación viral (Sallard et al., 2020). El tratamiento a base de interferones tipo I se ha investigado utilizándolos en combinación con Vilona® (ribavirina) o con la dupla lopinavir/ritonavir (Hung et al., 2020; Sanders et al., 2020; Sheahan et al., 2020). Estos estudios tratan de precisar si los efectos de este tipo de tratamiento combinado se deben solo al interferón, a la combinación de fármacos o a un efecto sinérgico.
Los interferones tipo I tienen diferentes grados de actividad antiviral, por ejemplo, el IFNβ parece tener mayor actividad en contra del coronavirus en comparación con el IFNα (Lam et al., 2020, Dong et al., 2020), que sería el tratamiento adecuado para probar en las etapas iniciales del COVID-19. Como se puede apreciar, los interferones, solos o en combinación con fármacos antivirales, ofrecen una interesante línea de investigación que debe proseguir ensayos clínicos exhaustivos con la finalidad de determinar la posibilidad de un tratamiento efectivo.
Uso de plasma de pacientes convalecientes de COVID-19
El plasma de pacientes convalecientes se ha utilizado para mejorar la tasa de supervivencia de los afectados durante los brotes del SARS-CoV (Yeh et al., 2005), el MERS-CoV (Arabi et al., 2016) o el virus del ébola (Marano et al., 2016), razón por la cual se ha retomado la idea de un tratamiento a base de plasma de pacientes que se han recuperado del COVID-19, esperando que su plasma contenga anticuerpos que puedan ayudar a enfrentar la enfermedad (Casadevall et al., 2020). Los primeros estudios al respecto tienen algunos resultados positivos (Shen et al., 2020; Cao et al., 2020b) y la FDA ha proporcionado recomendaciones detalladas para el uso de plasma convaleciente COVID-19 en investigación (FDA, 2020c). Este tipo de tratamiento sería beneficioso pensando en una profilaxis posterior a una exposición, especialmente en caso del personal sanitario que está constantemente en alto riesgo (Yeh et al., 2020). En este orden de ideas, se han registrado más de 95 ensayos en el clinicaltrials.gov (Keni et al., 2020), siendo elegibles para la donación las personas menores de 67 años, con una prueba de laboratorio positiva confirmada previamente para el COVID-19, estar libres de síntomas y completamente recuperado del virus durante al menos 28 días. Los ensayos clínicos están dirigidos a determinar una reducción consistente de la viremia, con incremento en la respuesta inmunológica y, se presta una especial atención a la reducción de la tormenta de citoquinas (Brown & McCullough, 2020). Los resultados iniciales encontrados en el uso de plasma convaleciente COVID-19 indican una reducción significativa de la carga viral con remisión de los signos y síntomas y disminución de la mortalidad (Rajendran et al., 2020). Por otro lado, el uso de esta metodología podría apuntar hacia la investigación con anticuerpos monoclonales (Marovich et al., 2020) dirigidos a inhibir la unión del virus al receptor ACE-2, una estrategia clave en el tratamiento contra el SARS-CoV-2.
Azitromicina, corticosteroides y otros agentes coadyuvantes en el tratamiento del COVID-19
La azitromicina (AZI) (Firth & Prathapan, 2020) mostrada en la figura 6, es un antibiótico de uso extendido contra bacterias grampositivas y gramnegativas, se ha utilizado en el tratamiento del COVID-19, y se hace asociada a hidroxicloroquina. De hecho, se ha sintetizado una sustancia híbrida entre azitromicina e hidroxicloroquina, un compuesto de la serie de las 4-amino-7-cloroquinolinas, que tienen una alta selectividad hacia Plasmodium falciparum, y su actividad antipalúdica in vitro es 1.000 veces mayor que la azitromicina sola (Pešić et al., 2012). Uno de los ensayos clínicos sobre el tratamiento con la mezcla azitromicina/cloroquina, administrada individualmente o en combinación, realizado en 1.438 pacientes COVID-19 (Rosenberg et al., 2020), no mostró diferencias significativas entre el grupo experimental y el control. A partir de la evidencia encontrada se concluye en la necesidad de realizar un mayor número de ensayos clínicos que permitan determinar la relación riesgo/beneficio, especialmente en lo que se refiere a la seguridad y eficacia en la asociación de cloroquina, hidroxicloroquina y azitromicina, para el tratamiento de pacientes afectados por COVID-19.
Medrol® (metilprednisolona) (Furman, 2019) (figura 6) y los corticosteroides en general, se consideran como adyuvantes en el tratamiento del COVID-19, puesto que se han utilizado ampliamente para tratar la neumonía grave y prevenir el daño pulmonar debido a su capacidad para suprimir la inflamación sistémica grave (Meduri et al., 2007). Existen varios estudios y revisiones sistemáticas de neumonías virales, incluidas SARS-CoV y MERS-CoV, donde se han aplicado tratamientos a base de corticosteroides, pero la evidencia clínica no es concluyente (Li et al., 2020b, Liu et al., 2020b; Russell et al., 2020a). Además, la administración temprana de corticosteroides, especialmente en dosis altas, podría llegar a ser perjudicial (Zhou et al., 2020b), pero en dosis bajas pueden ser terapéutico para los pacientes con un cuadro grave de COVID-19 y en general enfermos del síndrome de dificultad respiratoria aguda (SDRA), con sepsis o shock séptico (Zhou et al., 2020c). La falta de resultados concluyentes (Yang et al., 2020) indican que el uso de corticosteroides para el SARS-CoV-2 debe continuar la ruta de los ensayos clínicos exhaustivos (Russell et al., 2020b) pero, en todo caso, un tratamiento de esta naturaleza debe ser evaluado individualmente y considerar la gravedad de los síntomas, el momento de la intervención, la duración del tratamiento y la dosis a ser administrada.
Foipan® (mesilato de camostat) (Hsieh & Hsu, 2007) (figura 6), medicamento desarrollado en la década de 1980 para el tratamiento de la pancreatitis (Yamawaki et al., 2018), dispepsia (Ashizawa et al., 2006) hematuria y/o proteinuria (Asami et al., 2004) y más recientemente utilizado en MERS-CoV (Shirato et al, 2013), ha sido considerado como una posibilidad en los estudios sobre el COVID-19, debido a que se comporta como un eficiente inhibidor de proteasa contra tripsina, plasmina, calicreína, trombina, serina y de la TMPRSS2, esta última de gran importancia para la investigación en el 2019-nCoV (Saul & Einav, 2020; Luan et al., 2020; Gil et al., 2020; Hoffmann et al., 2020).
Sobre la base del conocimiento del mecanismo de infección de SARS-CoV-2, se sabe que la enzima convertidora de angiotensina 2 (ACE2) es un receptor celular expresado en arterias, corazón, riñones y pulmones, que se une a la proteína S, la espiga viral (figura 1), y constituye el receptor de entrada celular para el nuevo coronavirus. La proteína S se divide en dos subunidades, S1 y S2, la primera se une a ACE2, y la segunda, se divide y se activa a través de la proteasa transmembrana de serina 2 asociada a la superficie del hospedero, denominada TMPRSS2. Estas dos acciones generan como resultado una fusión de la membrana viral con la del hospedero, momento en el cual ARN viral se libera en el citoplasma de la célula y comienza la replicación del 2019-nCoV. En consecuencia, el mesilato de camostat es un agente farmacológico potencial para inhibir la entrada del SARS-CoV-2 en las células, previniendo la infección inicial (Uno, 2020). Sobre la base de esta información se han realizado varios ensayos, pero es necesario continuar la investigación a fin de establecer un posible tratamiento no solo en la vía de inhibición de la proteasa TMPRSS2 (Singh et al., 2020; Huggins, 2020; Kumar et al., 2020; Bittmann et al., 2020, Shrimp et al., 2020), sino en el área de los receptores de la enzima convertidora de angiotensina 2 (ACE2) (Ragia & Manolopoulos, 2020; Albini et al., 2020; Wu et al., 2020c; Cannalire et al., 2020; Teng & Tang, 2020).
La vacuna contra el COVID-19
La pandemia del nuevo coronavirus atrajo la mirada de opinión pública sobre la comunidad científica internacional que recibió una tarea tripartita, por una parte, estudiar en detalle el virus 2019-nCoV y su patogenicidad; en segundo lugar, encontrar tratamientos eficientes para tratar el COVID-2019 y, finalmente, encontrar una vacuna eficaz y segura que permita controlar el SARS-CoV-2, que ofrezca a la humanidad una oportunidad para enfrentar este desafío biológico (Kaur & Gupta, 2020). Muchos laboratorios y empresas se dieron a la tarea de desarrollar rápidamente posibles vacunas (Graham, 2020) y, en tal sentido, existen registrados 160 ensayos de los cuales muy pocos han entrado en fase I, II y III en los últimos 6 meses (Jeyanathan et al., 2020; Mahase, 2020).
No obstante, la imperiosa necesidad de la vacuna para el COVID-19, es necesario tomar en cuenta todas las consideraciones éticas o, mejor, bioéticas (Contreras, 2005), que giran alrededor de semejante esfuerzo científico mundial, pues se trata no solo de la necesidad de alcanzar a desarrollar la vacuna en un tiempo muy corto (Schaefer et al., 2020), sino que se está haciendo un ensayo clínico en seres humanos, tanto sanos como enfermos (Eyal, 2020).
Está claro que el objetivo de obtener una vacuna contra el COVID-19 es proteger a las personas antes de que se expongan al virus y, en la búsqueda de alcanzar la eliminación viral, induciendo en la persona vacunada una respuesta inmune, se está produciendo un cambio en el equilibrio inmunológico de su organismo y, una vez superado este proceso, debe alcanzar de nuevo el equilibrio sin producir ningún daño. En este sentido, se han establecido una serie de principios que se deben cumplir para obtener una vacuna segura y efectiva contra el COVID-19 (Thames et al., 2020), los cuales toman en cuenta la importancia de la especificidad de los anticuerpos humorales y la respuesta de las células T (Chen & Wherry, 2020). Hasta el momento, las investigaciones publicadas sobre el MERS y SARS aportan información valiosa (Al-Kassmy et al., 2020), entre otras que el receptor clave es la enzima convertidora de angiotensina 2 humana (ACE2). Adicionalmente, se conoce que están disponibles varias estrategias para el desarrollo de vacunas (Caddy, 2020), que toman en cuenta, entre otros aspectos, los vectores del virus, subunidades de proteínas, vacunas genéticas, anticuerpos monoclonales para inmunización, pero cada una de estas estrategias tiene su propio balance riesgo/beneficio (Hotez et al., 2020).
Parece estar claro que la carrera por la vacuna está comenzando (Lurie et al., 2020), pues no solo se trata de superar las tres etapas del ensayo clínico, sino que una vez alcanzada una vacuna viable y segura, comienza un complicado proceso de vacunación que debe ser equitativo, tomando en cuenta que se deben producir millones de dósis que deben estar disponibles para todos los países sin que se imponga condiciones (Bollyky et al., 2020). El desarrollo de una vacuna contra el COVID-19, dadas las circunstancias, impone un reto no solo técnico y operativo (Corey et al., 2020), sino desde el punto de vista ético (Meagher et al., 2020; Matsui et al., 2020), tomando en cuenta no solo la naturaleza misma de la investigación sino las decisiones que se deben tomar respecto de quienes serán los segmentos de la población que tendrán prioridad en el proceso de vacunación. Por ejemplo, los criterios de vacunación deben entrar a considerar los individuos de alto riesgo mayores de 60 años, particularmente aquellos con patologías previas, también se deben tomar en cuenta los trabajadores de la salud, especialmente los que brindan atención médica de primera línea (Chou et al., 2020) y, así mismo, todo el personal involucrado en industrias esenciales. Por otra parte, es menester tomar en cuenta aquellas personas que se han recuperado del COVID-19, pero que han desarrollado una inmunidad deficiente y por lo tanto pueden volver a contagiarse. Al final, cualquiera que sea el criterio seleccionado, siempre existirán motivos para objetarlos o discutirlos.
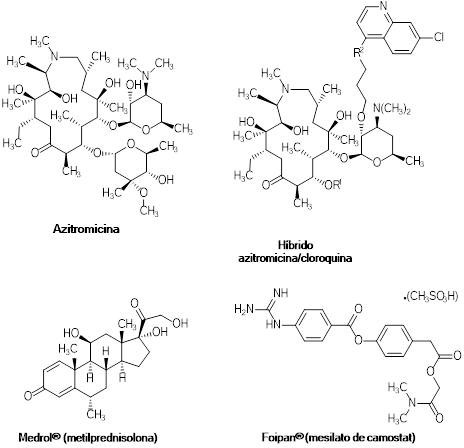
Figura 6: Estructura química de los fármacos: Azitromicina (AZI) (Firth & Prathapan, 2020); el híbrido azitromicina/cloroquina (Pešić et al., 2012); Medrol® (metilprednisolona) y Foipan® (mesilato de camostat) (Hsieh & Hsu, 2007).
Conclusiones
La pandemia del COVID-19 representa un desafío global que requiere una respuesta rápida, pero a la vez eficiente y eficaz en varios aspectos, entre ellos el desarrollo de nuevos tratamientos, y los correspondientes ensayos clínicos estandarizados con altos criterios bioéticos. Las tasas de infección por COVID-19 están en constante evolución, y es difícil predecir el comportamiento de una enfermedad frente a una sociedad donde los cambios económicos, políticos y socioculturales se producen a una velocidad sin precedentes. Sin embargo, el sector científico está respondiendo en esta carrera contra reloj frente al nuevo coronavirus 2019-nCoV, y se están produciendo importantes avances en la investigación sobre la producción agentes farmacológicos de diverso tipo, incluyendo antivirales, agentes inmunomoduladores, fármacos coadyuvantes y, así mismo, se están dando avances importantes en el desarrollo de posibles vacunas. En la actualidad, los esfuerzos también van en la dirección de producir un diagnóstico rápido y confiable, y en hacer un seguimiento y acompañamiento de los pacientes COVID-19 con estrategias dirigidas a tratar los síntomas y prevenir el desarrollo de infecciones graves. Se dispone del antiviral Veklury® o remdesivir, aprobado para tratar el SARS-CoV-2, pero existen muchos otros fármacos que tienen ensayos clínicos avanzados. En todo caso, la prevención sigue siendo el método de lucha contra el COVID-19, una enfermedad que la humanidad debe aprender a enfrentar pues se trata de una microscópica pero poderosa fuerza de la Naturaleza.