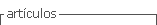Introducción
En la actualidad, la inminente pérdida de los recursos marinos naturales lleva consigo el desarrollo de la acuicultura de diversas especies, con la finalidad de cubrir el requerimiento de estos productos en el mercado, ya que existe una demanda insatisfecha de los productos del medio natural. Diversas especies de peces pueden cultivarse en diferentes ambientes, como estanques de barro, cemento, hapas y jaulas (Montenegro y Vallejo, 2015).
Una especie con potencial acuícola en desarrollo es el chame (Dormitator latifrons), un pez cuya distribución natural va desde California (Estados Unidos de Norte América), hasta las costas de Perú. Esta especie, es cultivada a mediana escala en Ecuador, y existe interés en México y Nicaragua para desarrollar su producción a través de la acuicultura. Como antecedente relevante, en Ecuador la producción total en cultivo esta reportada en el orden de 2000 toneladas por año hasta 1993, decayendo hasta 50 toneladas en 1997, con un leve repunte en años recientes de 800-1000 toneladas por año (Rodríguez et al., 2012). El chame es un pez tropical que pertenece a la familia Eleotridae. Su ciclo de vida se inicia cuando los adultos migran a aguas salobres para su reproducción, este período se da mayormente en época de lluvias o húmeda (Zambrano, 2014).
El chame es una especie con potencial para la acuicultura en Latinoamérica. Sin embargo, su producción depende de la utilización de individuos juveniles capturados en el medio silvestre, siendo necesaria la reproducción en cautiverio (Rodríguez et al., 2012). Las larvas de esta especie pueden iniciar la alimentación exógena al cuarto día después de la eclosión, después de consumir toda la yema de reserva, y cuando el tracto digestivo y los órganos asociados se han desarrollado por completo (López-López et al., 2015).
Este producto acuícola es muy atractivo en la gastronomía de Ecuador y despierta interés a nivel mundial debido a su sabor exquisito, y su importancia en la gastronomía de muchos países. Actualmente su producción en acuicultura, depende de la obtención de alevines del medio natural. Ante esta circunstancia, surge la necesidad de avanzar en la investigación de la eco biología de esta especie, para lograr producirla en ciclo cerrado y con ello generar dietas adecuadas para las diferentes etapas de su ciclo de vida.
Por la dependencia de juveniles del medio natural para la acuacultura del chame, se han desarrollado experimentos que tratan de permitir su reproducción y producción en ciclo cerrado, uno de estos experimentos es la inducción con hormonas estimuladora de la gonadotropina (LHRHa, GnRHa de salmón y domperidona), logrando exitosamente la liberación de esperma entre las 48 y 72 horas post-inyección (Rodríguez et al., 2012). Además, se ha realizado la inducción hormonal de reproductores y posterior fecundación artificial, logrando obtener prolarvas viables. Se evaluó el comportamiento de las prolarvas a lo largo de los días. Las larvas al cuarto día de post eclosión habían absorbido casi en su totalidad el saco vitelino y su cavidad bucal estaba abierta, listas para la alimentación exógena. Se trató de nutrir a las larvas con alimento natural, pero no se alimentaron y murieron (López et al., 2015).
Cedeño (2013), evaluó los efectos de la alimentación de chame cultivado en estanques de tierra con algas (del mismo sustrato natural del que se obtuvieron los peces), estiércol bovino secado al sol y alimento balanceado comercial. Los mejores valores de crecimiento se obtuvieron con el alimento balanceado, pero el mejor desempeño (costo/ganancia de peso) se obtuvo con estiércol bovino. Desafortunadamente, el autor no analizó las razones de la efectividad del estiércol bovino, ni su valor nutricional; por lo que posiblemente su contribución más significativa fue como fertilizante del sustrato del estanque, más que como un alimento altamente nutritivo.
Por su parte, Agualsaca (2015), evaluó el crecimiento de chame en cultivos de 46 días, utilizando detritos naturales y alimento balanceado con 32% de proteína. El autor informó, que los peces alimentados con detritos crecieron más lentamente que los alimentados con balanceado, utilizando raciones de alimento equivalentes al 4% de la biomasa total; lo que sugiere que los detritos, aunque forman parte de la dieta natural de D. latifrons, no aporta todos los nutrientes necesarios para su óptimo desarrollo.
En cuanto a los requerimientos nutricionales de D. latifrons, ningún estudio ha producido suficiente información para determinar que dieta se debe utilizar para esta especie. Se localizó una investigación sobre el efecto de varias concentraciones de proteínas y lípidos, en el crecimiento de ejemplares juveniles mantenidos en cautiverio, donde concluyó que una dieta con un 30% de proteínas y un 8% de lípidos era suficiente para asegurar un buen desarrollo de estos peces (Badillo et al., 2018).
Recientemente, Badillo et al. (2021), experimentaron con la sustitución parcial y total de harina de pescado por harina de soya, en la alimentación de juveniles; los resultados no mostraron efectos negativos en el crecimiento y la digestibilidad aparente, lo que sugiere que la especie absorbe una cantidad adecuada de nutrientes de fuentes vegetales.
Ante tal problemática la proteómica, un campo de la ciencia post-genómica, puede contribuir a dilucidar las redes funcionales de los sistemas biológicos, mediante un análisis directo de las proteínas expresadas. De esta manera, la proteómica aparece como un área de investigación que permite estudiar la dinámica de la expresión génica, que acompaña a múltiples procesos celulares (incluyendo desórdenes biológicos) manifestados en la localización, modificación e interacciones entre proteínas (Guajardo, 2005).
La proteómica es una herramienta poderosa para monitorear la dinámica biológica a niveles de proteína. Tiene aplicaciones beneficiosas en la ecología del agua, para la determinación de productos químicos industriales, como los estabilizadores UV en diferentes afluentes y efluentes ambientales (Liu et al., 2014), e incluso en lodos de agua (Casado et al., 2013). Además, monitorea el contenido de proteínas de los tejidos, mediante la identificación de las mismas y su abundancia diferencial durante las condiciones de desarrollo, alimentación, estrés y enfermedad (Forné et al., 2010), lo que proporciona una comprensión holística de los eventos moleculares y los mecanismos de las funciones del organismo (Li et al., 2016b).
Durante la última década también se ha observado una tendencia creciente a utilizar estas tecnologías ómicas en la acuicultura. Con el avance de las técnicas proteómicas, se pueden abordar muchos problemas de la acuicultura relacionados con el crecimiento, la resistencia a las enfermedades, la seguridad y la calidad de los alimentos acuáticos y los desafíos relacionados con el cambio climático (Nissa et al., 2021).
La tendencia a la degradación y la complejidad de las proteínas durante el procesado de las muestras, requiere desarrollar el método más adecuado para obtener tipos de proteínas de alta calidad, a partir del tejido vivo de las especies estudiadas. El objetivo de esta investigación es evaluar dos protocolos de extracción de proteínas, a partir de alevines de Dormitator latifrons. Una vez extraídas las proteínas, se comparó la concentración de proteína total y el patrón proteico obtenido con cada método, con el objetivo de designar el método más idóneo a utilizarse en estudios proteómicos con esta especie.
La investigación constituye una contribución a la acuicultura del chame. Este conocimiento resulta fundamental para la obtención de muestras proteicas que serán sometidas posteriormente a análisis de espectrometría de masas, lo cual a su vez posibilitará la identificación y cuantificación de las proteínas presentes en los alevines de chame durante sus primeras etapas de desarrollo. Este logro tiene el potencial de optimizar las fórmulas dietéticas que serán administradas a estos peces en sus etapas iniciales de vida.
Metodología
Se desarrolló un estudio experimental de laboratorio, con un enfoque en la comparación de dos métodos de extracción de proteínas y el análisis de los perfiles proteicos resultantes.
Muestreo, obtención, y conservación de los alevines
Para la realización del presente trabajo, se capturaron del medio natural 10 alevines y una prolarva de Dormitator latifrons. Los muestreos se realizaron en la parroquia Charapotó, Provincia de Manabí (Ecuador). Una vez capturados, fueron lavados con agua destilada y se realizó su inmersión en N2 líquido de forma inmediata; posteriormente se colocaron individualmente en tubos (Eppendorf) de 2 mL y fueron conservados en ultra congelación a -80°C hasta su utilización.
Preparación de soluciones buffers (tampones) para extracción de proteínas
En esta investigación se compara la efectividad de dos tampones de extracción de proteínas. Por un lado, un tampón de extracción compuesto por bicarbonato de amonio 50 mM (BioUltra, ≥ 99,5%); y por otro, el buffer RIPA, cuya composición se describe en la tabla 1:
Extracción de proteínas
La extracción de proteínas se realizó en el Laboratorio de Biología Molecular de la Universidad Técnica de Manabí (UTM), todo el proceso se realizó en frío. Cada alevín se cortó longitudinalmente y se hicieron dos alícuotas, de aproximadamente 500 mg cada una, que fueron conservadas en tubos de 2 mL (una alícuota de cada mitad para cada tampón de extracción a utilizar). A continuación, se agregó 1 mL de cada tampón (RIPA o bicarbonato amónico) en el tubo correspondiente.
Las muestras fueron homogeneizadas con maceradores plásticos de AXYGEN. Luego se centrifugaron a una velocidad de 14.000 g, a 4°C durante 15 minutos. Después de centrifugar, de cada tubo se extrajeron 2 alícuotas de 450 L del sobrenadante colocándolas respectivamente en 2 tubos Eppendorf de 1,5 mL, que se rotularon de acuerdo al tampón utilizado. La prolarva, debido a su pequeño tamaño, sólo se procesó con bicarbonato amónico. Una vez finalizado el proceso con todos los tubos, se almacenaron a -80°C.
Cuantificación de proteína total
La cuantificación de proteínas se realizó en uno de los laboratorios del Departamento de Bioquímica y Biología Molecular de la Universidad de Córdoba (España), utilizando el método de Bradford (1976). Para ello se construyó una recta de calibrado utilizando como proteína estándar la albúmina de suero bovino (BSA).
Cada una de las diluciones realizadas de las muestras, así como cada punto de la recta de calibrado, se midieron por duplicado en una placa de 96 pocillos. Para ello, se mezclaron 100 μL de cada muestra con 150 μL de una dilución 1:2 del reactivo de Bradford (Pierce) y se dejó incubar a temperatura ambiente durante 15 minutos, antes de medir la absorbancia a 595 nm (nanómetros). Para la cuantificación se empleó un espectrofotómetro para microplacas Multiskan GO y los resultados se analizaron mediante el programa SkanIt RE integrado en el Multiskan GO.
Electroforesis de proteínas en condiciones desnaturalizantes (SDS-PAGE)
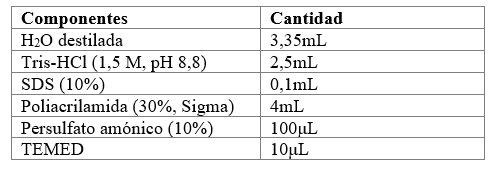
Para determinar cuál técnica de extracción de proteínas es más eficiente, se realizó un estudio del perfil proteico de cada muestra, mediante electroforesis en gel de acrilamida en condiciones desnaturalizantes (Laemmli, 1970). Para ello, se utilizaron geles discontinuos formados por un gel concentrador y un gel separador, las composiciones de los geles se describen en las tablas 2 y tabla 3. El gel concentrador, propicia que las proteínas ingresen todas al mismo tiempo en el gel separador.
Las muestras fueron tratadas antes de la electroforesis, para la desnaturalización de proteínas, mezclándolas al 50% con el tampón de carga Laemmli. A continuación, fueron incubadas 5 minutos a 95°C. En el gel se depositaron 3 μL del marcador de peso molecular (BioRad) y 30 μL de cada muestra. La electroforesis se llevó a cabo aplicando un voltaje de 100 V (voltios), hasta que las muestras alcanzaron el gel separador y a un voltaje de 200 V durante el resto de la electroforesis. Las proteínas se visualizaron mediante su tinción en una solución de azul Coomassie.
Análisis de resultados
Se utilizó el programa Microsoft Office\root\Office16\EXCEL.EXE, con el cual se realizó un gráfico de barras y desviación estándar muestral, para comparar las cantidades de proteínas obtenidas (método de Bradford) en cada muestra, de la extracción realizada con los dos buffers (RIPA y bicarbonato amónico).
Resultados
Cuantificación de proteína total
En el trabajo se han comparado dos métodos de extracción de proteínas de alevines de chame. Uno de los métodos lo compone el tampón RIPA utilizado de forma tradicional, aunque formado por compuestos no compatibles con espectrometría de masas. El tampón bicarbonato amónico, sin embargo, si es compatible con espectrometría de masas. Tras la extracción con los dos tampones, se procedió a estimar la eficiencia de la extracción mediante la cuantificación de proteína total.
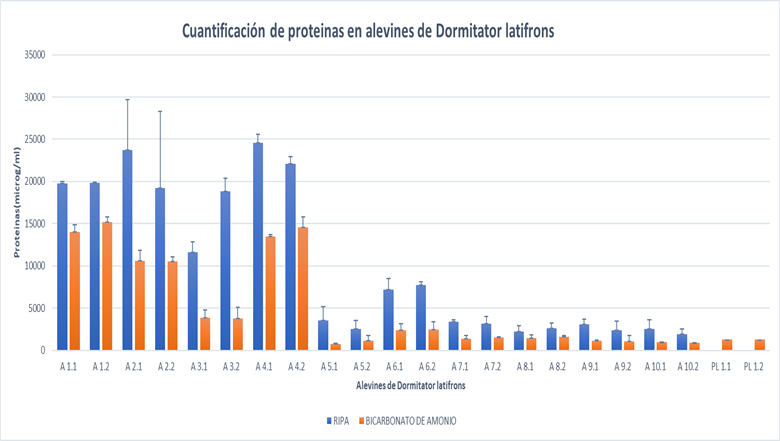
Al observar los resultados obtenidos con el método de Bradford, se evidenció que existen diferencias en la concentración de proteína total entre las muestras procesadas con los dos tampones de extracción: RIPA y bicarbonato de amonio respectivamente. Cabe mencionar que en cada proceso de extracción se trabajó con la misma muestra, ya que el alevín fue dividido en dos partes iguales, cada una para cada técnica y adicionalmente con cada método se generaron 2 alícuotas de extracto proteico.
En todas las muestras procesadas, se observa que la concentración de proteína obtenida, es mayor en todas aquellas en que se realizó la extracción de proteínas con el buffer RIPA en relación con el uso de bicarbonato amónico, como se muestra en la figura 1, a todas las muestras se les realizó la desviación estándar muestral. Este escenario puede hacer pensar que la técnica con RIPA es más recomendable, sin embargo, esto no garantiza que sea la más adecuada para trabajar en espectrometría de masas debido a su composición, tal y como se ha comentado anteriormente.
Análisis del perfil proteómico
Por otro lado, se evaluó el perfil proteico de las distintas muestras para valorar la complejidad de los extractos obtenidos con los dos tampones. Los resultados obtenidos mediante SDS-PAGE, muestran que ambos métodos de extracción son reproducibles (Figura 2), ya que los patrones obtenidos con las réplicas son muy parecidos si comparamos muestras extraídas con el mismo método.
Sin embargo, llama la atención la gran diferencia existente entre los perfiles obtenidos con ambos métodos de extracción. En las muestras procesadas con bicarbonato de amonio (C) (figura 2) se observa una mayor complejidad, con proteínas de tamaños variables a lo largo del intervalo de tamaño analizado; con este método hay una banda de alrededor de 40 kDa que destaca con por su mayor intensidad de coloración.
Las muestras tratadas con RIPA (R) que aparecen en la figura 2, se aprecia una banda es mucho más débil; en la extracción con RIPA se puede observar un perfil menos complejo y una mayor concentración de proteínas en la zona inferior del gel, zona donde se encontrarían las proteínas de menor tamaño. Estos resultados podrían indicar que en presencia del tampón RIPA las proteínas se degradan con mayor facilidad. De hecho, en las muestras extraídas con el tampón RIPA, puede observarse una franja del gel de coloración mucho más intensa en la zona cercana al frente, que podría corresponderse con péptidos obtenidos por degradación de proteínas en estas muestras.
Discusión
Los peces teleósteos, en su mayoría, pasan por una etapa de poco o nulo conocimiento de sus requerimientos proteicos y en especial las etapas tempranas de su vida, siendo esto el principal factor limitante para su acuicultura. El chame (Dormitator latifrons) tampoco es la excepción a esta problemática, dependiendo de semilla del medio silvestre para la práctica de la acuicultura, esto se corrobora con lo expuesto por Rodríguez et al. (2012). Su producción depende de la utilización de individuos juveniles capturados del medio silvestre, siendo necesaria la reproducción en cautiverio (López et al., 2015).
Se desconoce los porcentajes de proteínas necesarios para los juveniles de esta especie (Badillo et al., 2018). Las proteínas son las biomoléculas más abundantes, representando en varias especies, más del 50% del contenido total de nutrientes presentes en el vitelo (Abdo et al., 2017); representando la mayoría de los requerimientos nutricionales de los peces.
En el presente trabajo de investigación se ha identificado la técnica más idónea para la extracción de proteínas en alevines de chame, para su posterior análisis mediante proteómica cuantitativa. El objetivo a largo plazo de nuestro trabajo es identificar y cuantificar las proteínas necesarias a suministrar en las dietas de los alevines de esta especie, es decir después de la absorción del saco vitelino.
Se obtuvieron alevines del medio silvestre y fueron lavados en agua destilada, y sacrificados por inmersión en N2 líquido para luego ser almacenados a -80°C; esta técnica de bio criogenia se diferencia de la utilizada por Chicano et al. (2010), donde a los juveniles de lenguado senegalés (Solea senegalensis) se les realizó la inmersión en N2 líquido y para organismos adultos se efectuó la extracción de los órganos específicos a estudiar, y luego se realizó la inmersión de los mismos.
Para la extracción de proteínas, se utilizaron dos tampones (RIPA y Bicarbonato de Amonio) para comparar la eficiencia de estos. Chicano et al. (2010), utilizaron tampón TRIS y junto a estos cocteles inhibidores de proteasa, y benzonasa para evitar presencia de ADN o ARN en la muestra. En nuestro caso, con el objetivo de abaratar el proceso, no se les añadió ningún inhibidor de proteasas y para evitar la degradación de las proteínas se trabajó a temperaturas bajas.
En los resultados de la presente investigación se observó que al cuantificar las proteínas de las muestras con el método de Bradford (1976), se obtuvo una mayor concentración de proteína total en las muestras tratadas con el tampón RIPA. En base a este resultado, aparentemente, el tampón RIPA se mostraba como el más eficiente.
Dicha aseveración fue cambiada al analizar los perfiles proteicos en la SDS-PAGE; tras visualizar los patrones obtenidos mediante SDS-PAGE, se observó un perfil más complejo y definido en las muestras usando el bicarbonato de amonio en comparación con las de RIPA. Una de las causas podría deberse a que el tampón RIPA resulta de la combinación de varios reactivos, lo cual puede generar desechos residuales que interfieren en la electroforesis. Así mismo el tampón RIPA, aunque utilizado de manera tradicional, está constituido por compuestos no compatibles con espectrometría de masas; en cambio el tampón bicarbonato amónico, si es compatible con espectrometría de masas.
En otra investigación (Chicano et al., 2010), se utilizó un tampón similar al RIPA, este tampón fue el Tris-HCl 20 mM, pH 7,6, con sacarosa 0,5 M y KCl 0,15M; a razón de 2 ml/g de tejido; el tampón se suplementó con PMSF 1mM, leupeptina 0,006mM, y con un cóctel de inhibidores de proteasas, que contenía AEBSF, EDTA, bestatina, E-64, leupeptina y aprotinina; en esta investigación se realizó electroforesis bidimensional, en nuestro estudio utilizamos electroforesis en gel de acrilamida en condiciones desnaturalizantes, para determinar el perfil proteico de cada muestra.
Conclusiones
Los hallazgos de la investigación constituyen un paso de avance para el estudio del proteoma en las etapas tempranas del chame (Dormitator latifrons), permitiendo la optimización de la extracción de proteínas, que es sin duda un paso importante en la investigación proteómica de este pez de interés acuícola, concluyendo que el tampón más recomendado a utilizar es el bicarbonato de amonio y es el protocolo de extracción de proteínas más viable para proteómica, de entre los dos protocolos evaluados en esta investigación; toda vez el buffer RIPA, no mostró las condiciones específicas para su utilización.
En futuras investigaciones, se procederá a la identificación y cuantificación de las proteínas (espectrometría) en los alevines de chame, con el objetivo de mejorar la dieta de esta especie y así contribuir al avance de la acuicultura de este pez de alto valor nutritivo e interés económico.