Forma sugerida de citación:
Patiño, D.; Medina, J.; Silva, R.; Guijarro, A. y Rodríguez, J. “Predicción de arritmias e infartos agudos de miocardio usando aprendizaje automático,” Ingenius, Revista de Ciencia y Tecnología, N.◦ 29, pp. 79-89, 2023. DOI: https://doi.org/10.17163/ings.n29.2023.07
1. Introducción
Hoy en día se utiliza una multiplicidad de dispositivos (computadoras personales, teléfonos inteligentes, tabletas, teléfonos celulares, etc.) para acumular y procesar big data acerca del comportamiento humano. Esta base está disponible para una variedad de propósitos, incluyendo medicina [1, 2]. La salud móvil (mHealth, Mobile Health en inglés) y los dispositivos inteligentes permiten la detección temprana y la intervención inmediata en pacientes con fibrilación auricular (AF, Atrial Fibrillation en inglés). El ECG de una derivación y de múltiples derivaciones, la fotopletismografía (PPG, Photoplethysmography en inglés) y la medición oscilométrica con capacidades de diagnóstico validadas, pueden integrarse dentro de la práctica clínica para detectar AF. Las directrices existentes de práctica clínica sugieren que la evaluación del pulso con el examen ECG para la población de alto riesgo y para pacientes mayores a 65 años de edad, es apropiada para reducir las complicaciones. Sin embargo, los dispositivos de salud e inteligentes destinados al consumidor, fáciles de usar y económicos pueden ser una herramienta alternativa de examen no solo para la población mayor con comorbilidades, sino también para la población general de bajo riesgo con monitoreo frecuente [2,3,4,5,6,7].
Los dispositivos mHealth capaces de monitorear la frecuencia cardíaca y/o el ritmo cardíaco vienen en múltiples formas tales como apps para teléfonos inteligentes, relojes inteligentes, anillos, collares, sensores portátiles y parches [8,9,10]. Las compañías han creado productos capaces de generar un punto de cuidado a través de registros ECG, tales como la serie de dispositivos AliveCor Kardia Monitor [8]. En un estudio realizado en Hong Kong con un gran número de personas, la investigación sobre el dispositivo Kardia para ECG de una derivación encontró que un cardiólogo confirmó que 65 % de las AF detectadas por el dispositivo fueron precisas. En este estudio en más de diez mil pacientes con una edad promedio de 78 años, el número requerido para hacer nuevos diagnósticos de AF con precisión fue 145 participantes [8]. La sensibilidad y especificidad del monitor Kardia fueron 99.6 % y 97.8 %, respectivamente [11].
El término ‘salud ubicua’ (u-Health, Ubiquitous Health, en inglés), definido por Weiser como la integración de la computación en acciones y comportamientos humanos “en cualquier momento” y “en cualquier lugar” ha venido adquiriendo importancia [1, 2]. El principal atributo de u-Health es la capacidad de interacción entre individuos y dispositivos, de forma tal que la tecnología es transparente para el usuario [12]. No está claro cuál es el mejor algoritmo para detectar enfermedades cardiovasculares por medio de los dispositivos u-Health. Se requiere que la tecnología sea robusta, confiable y de bajo costo computacional, para que pueda ser ejecutada en los dispositivos, aun cuando están desconectados. El objetivo de este artículo es evaluar la mejor alternativa para la detección de arritmias con dispositivos u-Health.
Este trabajo es producto de una colaboración en curso entre la Universidad de Guayaquil y la Universidad de Villanova, bajo la cual se están desarrollando múltiples estrategias de inteligencia artificial para la detección de arritmias en tiempo real. El presente trabajo utiliza bases de datos existentes de arritmias para validar las estrategias, y el trabajo futuro intenta procesar data de dispositivos portátiles en tiempo real. Los resultados de esta investigación son altamente prometedores, y serán discutidos con mayor profundidad a lo largo del artículo.
2. Materiales y métodos
2.1. Metodología
Se utilizaron dos bases de datos Physionet de electrocardiogramas ECG, MIT (arritmias) con 109444 registros (21891 normales y 87553 anormales) y PTDB (infartos) con 14 550 registros (4045 normales y 10505 anormales). La base de datos MIT tiene cuatro categorías: Normal ‘N’ 0, Supraventricular ‘S’ 1, Ventricular ‘V’ 2, Fibrilación Ventricular ‘F’ 3, Otras no clasificadas ‘Q’ 4. La base de datos PTDB tiene 2 categorías: ‘N’ 0 y con problemas cardíacos ‘A’ 1. Se utilizó SMOTE para regularizar las categorías y evitar sobreajuste y subajuste.
El 80 % de los registros se utilizan para entrenamiento y el 20 % para prueba; el 20 % del 80 % del conjunto de entrenamiento se toma nuevamente para evaluar la predicción de los datos. Este proceso fue realizado por separado para las bases de datos MIT y PTDB, utilizando redes neuronales artificiales (ANN, artificial neural networks en inglés) y redes neuronales convolucionales (CNN, convolutional neural networks en inglés), además del algoritmo de decisión basado en árbol XGBoost o impulso del gradiente extremo; los tres algoritmos fueron evaluados con las dos bases de datos.
2.2. Enfermedades cardiovasculares e inteligencia artificial
Las enfermedades cardiovasculares (CVD, Cardiovascular Diseases en inglés) son la principal causa de mortalidad a nivel mundial, representando el 31 % de todas las muertes [13]. Una de las causas principales es el infarto agudo al miocardio (AMI, acute myocardial infarctation, en inglés). Existe una necesidad creciente de estudiar una amplia gama de técnicas de vanguardia para el análisis y diagnóstico de enfermedades cardíacas. Con el fin de evaluar la situación específica del paciente, los doctores frecuentemente analizan las señales del ECG (electrocardiógrafo) para obtener suficiente información que les ayude a hacer el diagnóstico. Muchos investigadores han aplicado algoritmos de aprendizaje automático (ML, Machine Learning, en inglés) para estudiar el problema de clasificación de arritmias [14]. Los avances en procesamiento de datos, capacidad de almacenamiento y métodos de aprendizaje automático han venido transformando el campo de la medicina, incluyendo la cardiología [15].
La AF es uno de los tipos más comunes de arritmia, que se caracteriza por un latido rápido e irregular [13]. La enfermedad cardíaca isquémica (IHD, Ischemic heart disease, en inglés) es una condición en la cual existe un suministro inadecuado de sangre y oxígeno a una parte del músculo cardíaco [16]. Esta condición ocurre usualmente cuando hay un desbalance entre el suministro de oxígeno y la demanda del músculo cardíaco (miocardio), debido típicamente a enfermedad cardíaca ateroesclerótica [17]. Usualmente los pacientes no muestran los signos y síntomas típicos (asintomático), hasta que la enfermedad cardíaca isquémica se manifiesta como angina, infarto al miocardio o muerte cardíaca repentina [18].
En el análisis de ECG, la clasificación de los latidos del corazón es la manera más común de automatizar el diagnóstico de la arritmia [19]. El flujo común de aprendizaje de ECG usando técnicas de aprendizaje automático incluye análisis señal-ruido, reconocimiento de los latidos del corazón, extracción de características y clasificación de los latidos del corazón. Para aprendizaje profundo, la extracción de características puede reemplazarse por el almacenamiento de fragmentos de latidos de una secuencia de ECG completa [20]. Los algoritmos reportados en la literatura para la identificación de señales de ECG incorporan tres puntos importantes: preprocesamiento, clasificación y desbalance del conjunto de datos. Para analizar las características que pueden estar directamente relacionadas a factores fisiológicos en el desarrollo de enfermedades, se consideraron tres algoritmos de aprendizaje profundo: redes neuronales convolucionales (CNN), redes neuronales artificiales (ANN) y el árbol reforzado de impulso de gradiente extremo o XGBoost.
Las redes neuronales artificiales son algoritmos de aprendizaje que pueden identificar relaciones complejas en los datos. Las ANN están diseñadas para imitar el sistema nervioso humano. Las ANN típicas están compuestas por tres tipos de capas: entrada, salida y ocultas. Cada capa está constituida por neuronas [21]. Las redes neuronales convolucionales (CNN), las redes neuronales recurrentes (RNN, recurrent neural networks en inglés) y el Naive Bayes se utilizan como clasificadores. Existen enfoques que combinas diferentes técnicas tales como Transformada Wavelet Discreta (DWT, Discrete Wavelet Transform, en inglés) y ANN para la clasificación de arritmias en ECG [22]. Cuando el número de características es mayor que el número de muestras, las ANN pueden manejar múltiples clases, no hay efecto de los grandes conjuntos sobre las ANN y no se requiere una memoria extensiva [23].
Un estudio basado en ANN clasifica IHD utilizando parámetros de variabilidad de la frecuencia cardíaca (HRV, heart rate variability en inglés) junto con una data clínica tal como la fracción de eyección del ventrículo izquierdo (LVEF, left ventricular ejection fraction en inglés), edad y género. Se probaron una serie de redes con diferente número de nodos de entrada (varía entre 7 y 15), nodos ocultos (entre 2 y 10) y dos nodos de salida. Los rangos de entrenamiento y prueba fueron 75 % y 25 %, respectivamente, del total de datos de entrenamiento [24]. Varios investigadores también han utilizado enfoques basados en redes neuronales artificiales para la clasificación diagnóstica de señales de ECG [16].
Las técnicas modernas de redes neuronales profundas (DNN, Deep Neural Networks en inglés) se utilizan para resolver el problema de selección y extracción manual de características en sistemas automáticos convencionales para el diagnóstico de imágenes de Infarto al Miocardio (MI, Myocardial Infartaction en inglés) [25]. El algoritmo de retropropagación se utiliza para entrenar redes neuronales profundas [26]. Las CNN se utilizan principalmente para analizar imágenes. El infarto al miocardio se predice utilizando imágenes características tomadas antes y después del ataque, como imágenes de entrada a una CNN [27]. Las capas comúnmente utilizadas en CNN son la convolucional (Conv), unidad lineal rectificada (ReLU, rectified linear unit, en inglés), pooling, normalización en lotes y capas totalmente conectadas [28]. Una matriz de entrada se alimenta a un modelo de detección constituido por CNN y una red de memoria larga de corto-plazo bidireccional (bi-LSTM, Long short-term memory network en inglés) con validación cruzada estratificada de cinco etapas [29].
Las DNN han mostrado ser exitosas en diferentes áreas, incluyendo imágenes, audio y texto [30]. En aplicaciones reales, el tipo de dato más común son los datos tabulados, que contienen muestras (filas) con el mismo conjunto de características (columnas). Los datos tabulados se utilizan en muchas áreas como medicina, finanzas, manufactura, ciencia climática y muchas otras [31].
Los métodos tradicionales de aprendizaje automático, tales como los árboles de decisión accionados por gradiente (GBDT, gradient-boosted decision trees en inglés) [32], dominan el modelado de datos tabulados y muestran un mejor desempeño que el aprendizaje profundo. A pesar de sus ventajas teóricas [33,34,35], las DNN plantean muchos retos cuando se aplican a datos tabulados, tales como falta de localidad, escasez de datos (valores faltantes), mezcla de diferentes tipos de características (numéricas, ordinales y categóricas) y falta de conocimiento previo acerca de la estructura del conjunto de datos (a diferencia del texto o las imágenes). La combinación de algoritmos de árboles, tal como el XGBoost, se consideran la opción recomendada para problemas reales involucren datos tabulados [32], [36].
Los XGBoost han sido utilizados para clasificar fibrilación auricular [37]. Un estudio propone un clasificador de señales de ECG basado en XGBoost y un conjunto de técnicas de descomposición empírica en modos (EEMD, ensemble empirical mode decomposition en inglés) que aprovecha funciones basadas en tiempo, frecuencia y características morfológicas [38].
Otro estudio propone crear un conjunto de características morfológicas en cinco dimensiones que incluyen complejos QRS e intervalos RR, así como también coeficientes de características wavelet, para construir el vector de características para una clasificación altamente eficiente de los latidos del corazón [21]. Las medidas de desempeño son entrenadas para encontrar características que sean clasificadas correctamente; entonces, la relación de aquellas que no son bien clasificadas se utiliza para encontrar la eficiencia del clasificador. Se puede obtener una proporción alta, aún si todas las clases importantes se clasifican de forma incorrecta. Para superar esto, la data debe estar adecuadamente balanceada [39]. La técnica de sobremuestreo de minorías sintéticas (SMOTE, Synthetic Minority Oversampling Technique, en inglés) puede superar algunas desventajas en la clasificación [40]. Este método ha mostrado ser mejor que otras mezclas de submuestreo y sobremuestreo.
Un estudio realizado en 2021 comparó el desempeño de un XGBoost y una DNN utilizando el optimizador Adamax y una función de pérdida de entropía cruzada con cuatro capas ocultas. Los resultados mostraron que el XGBoost tuvo un mejor desempeño que la DNN, ya que alcanzó una precisión de aprendizaje de 100 % y una precisión de predicción de 95.60 % y 93.08 % para las mismas fases [41]. El desempeño global de aprendizaje del modelo DNN fue 89.42 % y 81.23 %, mientras que su precisión en la predicción fue 80.50 % y 77.36 %, respectivamente, para las mismas variables [41]. El objetivo del presente estudio fue comparar los algoritmos para determinar la solución más económica para la detección de arritmias en tiempo real.
Los monitores de ECG de una sola derivación se utilizan frecuentemente debido a su naturaleza altamente productiva, corto tiempo de ejecución y bajo costo [42]. Sin embargo, los ECG de una derivación no pueden capturar toda la información debido a la gran diversidad de características de las CVD que pueden causar diagnósticos equivocados [43].
2.3. Inteligencia artificial
El objetivo de este trabajo es seleccionar un algoritmo para la clasificación de alteraciones cardíacas que pueda ser ejecutado en tiempo real, es decir, mientras se adquiere la señal electrocardiográfica. Considerando que los algoritmos evaluados se basan en la identificación y clasificación de un solo ciclo cardíaco, lo ideal sería tener un algoritmo capaz de capturar y clasificar las señales durante el período entre ondas, mejor conocido como segmento o intervalo T-P, como se muestra en la Figura 1.
Para ejecutar el algoritmo en tiempo real, el tiempo de ejecución debe ser menor que el intervalo T-P, es decir, menor a 200 ms. Se analizó la dispersión de los archivos de las bases de datos Physionet que están disponibles para investigación sobre electrocardiogramas (ECG). Dicha base de datos fue desarrollada por el Hospital Beth Israel en Boston (ahora el Beth Israel Deaconess Medical Center), juntamente con el Instituto de Tecnología de Massachussetts (MIT-BIH) [44] y el Physikalisch - Technische Bundesanstalt, the National Metrology Institute of Germany (PTB). La base de datos MIT-BIH tiene 109444 ECG y la base de datos PTB tiene 14550 ECG [45].
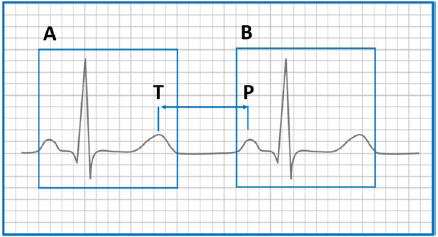
Figura 1. Dos Ciclos Cardíacos Independientes A y B dentro de su ventana de detección correspondiente con el intervalo T-P identificado
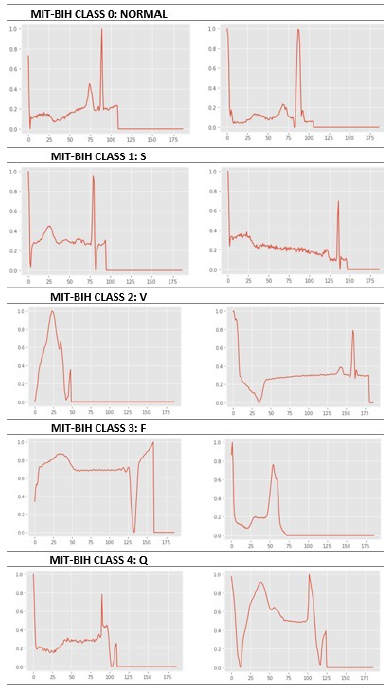
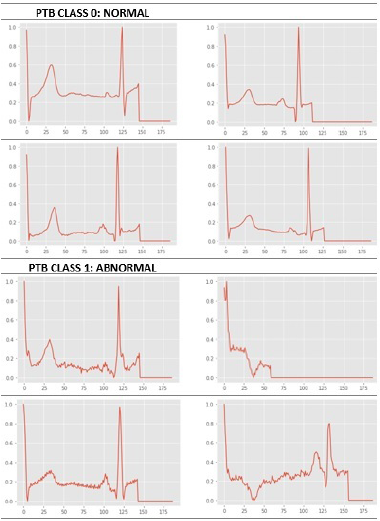
La MIT-BIH es una base de datos de arritmias, que tiene una clasificación etiquetada como ‘N’: 0, ‘S’: 1, ‘V’: 2, ‘F’: 3, ‘Q’: 4, donde 0 es NORMAL y de 1 a 4 son arritmias clasificadas como bradiarritmias y taquiarritmias, subclasificadas como taquiarritmias supraventriculares y ventriculares. En la base de datos PTB, la clasificación es 0 para NORMAL y 1 para ANORMAL, donde se consideran enfermedad cardíaca severa, tal como infarto al miocardio (principalmente), insuficiencia cardíaca y hemibloqueos. Los archivos csv disponibles en Kaggle tienen 187 columnas que representan la bioseñal del ECG y 188 columnas adicionales que clasifican el ECG. Este campo está disponible en ambas bases de datos y permite la aplicación de aprendizaje automático.
Se consideraron tres algoritmos: redes neuronales convolucionales (CNNs), redes neuronales artificiales (ANNs) y el árbol reforzado de impulso de gradiente extremo o XGBoost. Los trabajos publicados por Premanand S, disponibles en Analytics Vidhya, se tomaron como referencia para las redes neuronales.
Para evitar los problemas de subajuste y sobreajuste en aprendizaje automático, se aplicó la función SMOTE a ambas bases de datos por separado. Esto crea nuevos ECG sobre la base de la data original y balancea las categorías.
Se hizo una división por 80-20-20 a ambas bases de datos, obteniendo 289878-72470-90587 y 13446-3362- 4202 en datos de ECG para entrenamiento, prueba y validación en las bases de datos MIT y PTB, respectivamente. Las Figuras 2 y 3 incluyen gráficos de muestras de ECG de ambas bases de datos en las diferentes categorías normal y anormal.
Se entrenó utilizando los algoritmos ANN, CNN y XGBoost para las bases de datos MIT y PTB por separado, para hacer una comparación en términos de tiempos de entrenamiento y niveles de precisión en la predicción, es decir, exactitud, precisión y recall, generando las matrices de confusión correspondientes.
Los resultados del entrenamiento fueron inicialmente validados usando la data de validación y prueba de ambas bases de datos, MIT y PTB, por separado. Posteriormente, se valida el nivel de predicción cruzando datos de ambas bases de datos. Una arquitectura CNN propone seleccionar un grupo óptimo de capas individuales y el tamaño de los filtros. Se seleccionaron los siguientes valores: dos capas densas, tamaño de capa 128, número de capas convoluciones 2D y MaxPooling 2D [12 N] [46].
3. Resultados y discusión
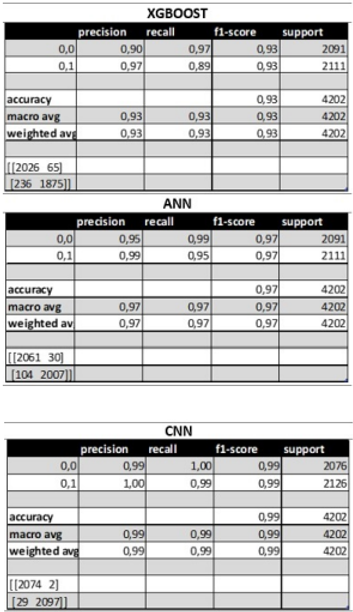
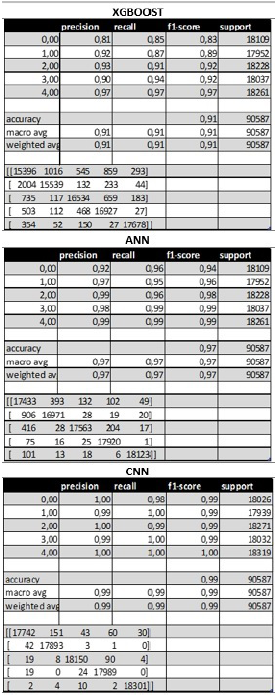
Se procedió a entrenar con ANN, CNN y XGBoost sobre las bases de datos MIT y PTB por separado, para establecer comparaciones en términos de tiempos de entrenamiento y niveles de precisión en la predicción: exactitud, precisión y recall. Las matrices de confusión correspondientes se presentan en las Figuras 4 y 5.
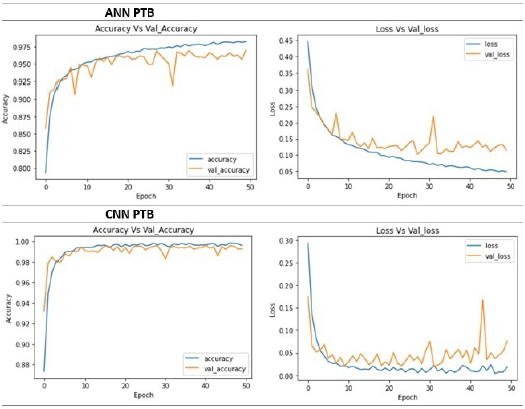
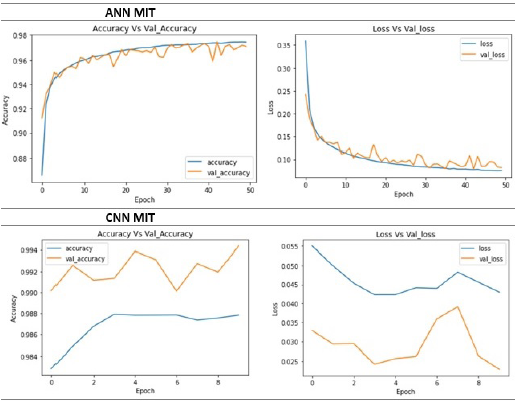
Las Figuras 6 y 7 presentan Exactitud vs. Pérdida para cada uno de los algoritmos, sobre las bases de datos PTB y MIT.
La Figura 8 presenta los tiempos FIT y la exactitud obtenidos para cada una de las bases de datos.
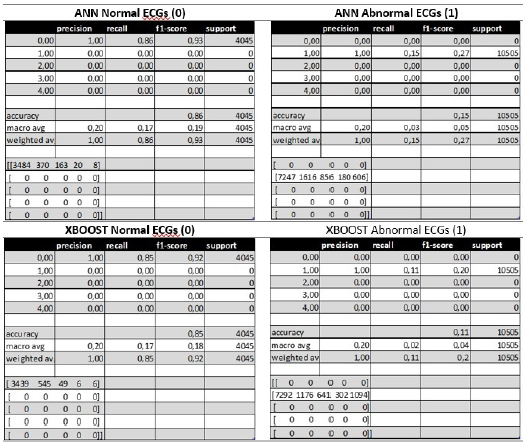
Luego de evaluar los resultados del entrenamiento por separado, se hizo una validación cruzada del nivel de predicción de los algoritmos intercambiando datos de ambas bases de datos, como se muestra en la Figura 9. Para propósitos de validación, la base de datos PTB fue categorizada como 0 = normal y 1 = anormal, y procesada por los modelos aprendizaje basados en XGBoost y ANN.
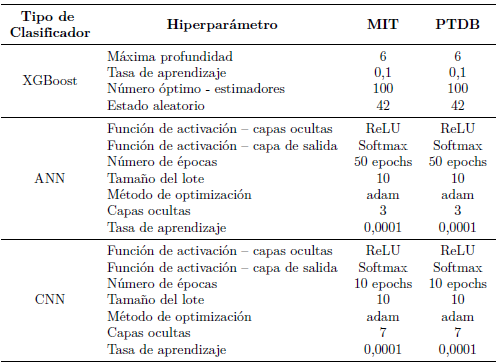
La fase de entrenamiento se completó a través de definiciones por ensayo y error; los hiperparámetros fueron configurados de acuerdo con la Tabla 1, para alcanzar el nivel de esperado de exactitud.
Se validó la predicción de los modelos de aprendizaje automático entrenados con los ECG de la base de datos MIT-BIH, sobre los ECG de la base de datos PTB, obteniendo niveles de exactitud de 85 % y 86 % para los ECG normales. Con respecto a la validación sobre las anormalidades, el XGBoost tuvo una exactitud de 11 % y la ANN tuvo 15 %. Esto se debe a que las bases de datos incluyen diferentes enfermedades cardíacas: arritmias en la MIT-BIH e infartos agudos al miocardio en la PTB.
Esto demuestra que las señales normales pueden ser reconocidas de manera cruzada entre bases de datos, pero la data correspondiente a ECG anormales no es interoperable entre una base de datos y otra.
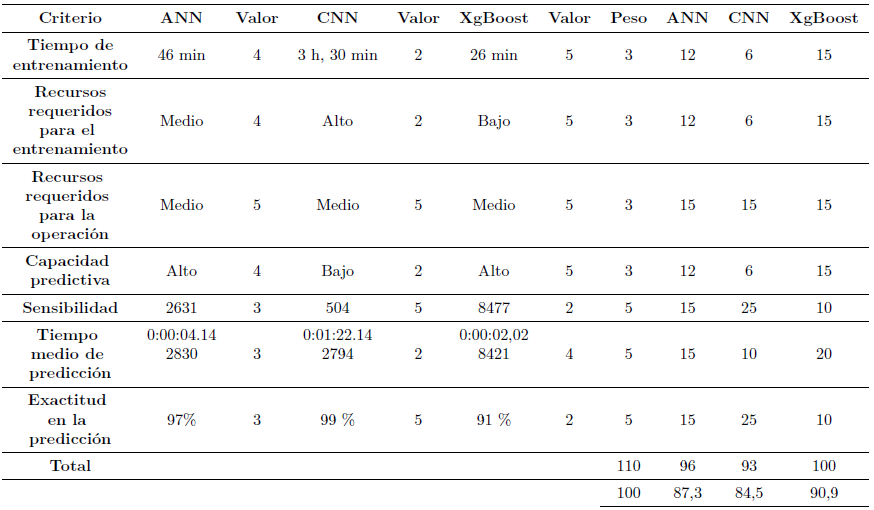
Finalmente, a través de aprendizaje supervisado, los dos modelos de redes neuronales artificiales y el algoritmo XGBoost para predicción por clasificación fueron comparados utilizando una matriz de pesos, considerando criterios tales como exactitud en la predicción, sensibilidad de la data médica (falso positivo/- falso negativo), tiempo de aprendizaje y tiempo de predicción, tal como se muestra en la Tabla 2.
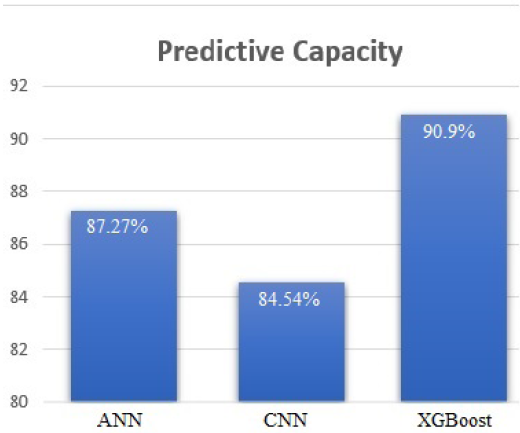
Como se definió previamente, el objetivo de este trabajo es seleccionar un algoritmo para la clasificación de alteraciones cardíacas que pueda ser ejecutado en tiempo real, es decir, mientras se están adquiriendo la señal electrocardiográfica. Dados los resultados de la Tabla 2, puede hacerse una comparación para determinar cuál es el mejor algoritmo para una solución u-Health (Figura 10).
Para la fase de aprendizaje de los modelos se utilizaron los 123 994 registros, y a partir de los valores pronosticados se determinó el modelo que alcanza la más alta exactitud de predicción. Considerando que la inteligencia artificial ha logrado aprender cómo clasificar apropiadamente condiciones asociadas a las patologías cardíacas, se seleccionó el modelo más adecuado y aplicable para el diagnóstico de gente o grupos poblacionales. En temas de prevención, la predicción del riesgo para la población en general es de vital importancia ya que puede reducir el impacto de las muertes debido a patologías cardíacas, así como también reducir los costos asociados a estos casos.
4. Conclusiones
A partir de la discusión, se usó una matriz de pesos para comparar la calidad de los tres algoritmos de predicción. Sobre la base de esos resultados, se concluye que las redes neuronales convolucionales (CNN) son mucho más precisas que los otros algoritmos (99 %); sin embargo, el tiempo de entrenamiento es largo (en términos de horas), comparado con el tiempo de entrenamiento del XGBoost que toma minutos. Dado que se está tratando con la salud humana, la precisión y exactitud en la predicción son más importantes que la rapidez de entrenamiento. En un punto intermedio está la red neuronal artificial (ANN), que alcanza un desempeño muy aceptable con una exactitud de 97 %.
Dada la naturaleza tabular de los datos, el XGBoost es la mejor alternativa, como se observa en la Figura 10.
La conclusión previa indica que es posible obtener información acerca de una arritmia dentro del intervalo RR. Dado que el objetivo del proyecto era procesar datos en tiempo real, los resultados son altamente prometedores. Como trabajo futuro, se pretende usar data de ECG generada por relojes inteligentes como parte de una investigación doctoral. La detección de arritmias a partir de relojes inteligentes sería una herramienta importante para la detección temprana de eventos potencialmente mortales, tales como fibrilación. Sin embargo, es necesario reducir los falsos positivos, y dado que se podría procesar la data en tiempo real, la distribución de probabilidad conjunta puede ser usada en trabajos futuros para aumentar la naturaleza predictiva del algoritmo. En resumen, esta es una contribución significativa en el campo de detección de arritmias en tiempo real.