1. Introducción
Los nematodos constituyen un importante problema de salud pública, generan potenciales riesgos de enfermedades infecciosas o zoonóticas, como las parasitosis.
Entre los parásitos intestinales más comunes que afectan a caninos y felinos domésticos y silvestres, se encuentran el Toxocara y Toxascaris, pudiendo causar un importante impacto en la salud pública, especialmente las especies de Toxocara [1, 2, 3].
Los humanos son huéspedes accidentales de Toxocara spp. y se infectan a través de la ingestión de huevos infecciosos del medio ambiente o de artículos contaminados con heces de animales infectados, como es el caso del contacto con áreas de tierra que los contienen. Luego, las larvas liberadas en el intestino migran hacia los tejidos, provocando síntomas clínicos que incluyen larva migrans visceral, ocular larva migrans, meningoencefalitis eosinofílica, toxocariasis encubierta y toxocariasis neurológica [3].
Las características morfológicas y las preferencias del hospedero han sido empleadas para la identificación de especies, pero estas claves y descripciones existentes en ocasiones son insuficientes para el diagnóstico, particularmente cuando se requiere diferenciación dentro y entre Toxocara spp. y Toxascaris spp., especialmente en las etapas de larva y huevo [1, 4,].
En particular, la detección de los parásitos nematodos como Toxocara canis, Toxocara cati y Toxocara leonina se realiza fundamentalmente a través de análisis microscópico y pruebas serológicas. Sin embargo, ambos métodos resultan insuficientes para diferenciar especies estrechamente relacionadas como lo son el T. canis y el T. cati.
El diagnóstico molecular es un método que se encuentra en continuo desarrollo, permitiendo la detección e identificación con precisión de los n nematodos ascaridoides. Las técnicas basadas en la reacción en cadena de la polimerasa (PCR), utilizando marcadores genéticos tanto del genoma nuclear, como mitocondrial, han evolucionado el diagnóstico parasitario hacia una mayor especificidad y rapidez. Como en el caso del segundo espaciador transcrito interno (ITS-2) utilizado para identificar nematodos en perros (Canis lupus familiaris), zorros (Vulpes vulpes) y gatos (Felis catus), permite la identificación precisa.
Por ello, la identificación del Toxocara canis de aislamientos en caninos del cantón Salcedo, provincia Cotopaxi (Ecuador), mediante la secuenciación de la región del espaciador transcrito interno (ITS-2) del ADN ribosómico (ADNr), constituye el objetivo del presente estudio.
2. Materiales y métodos
La investigación se realizó en los barrios del cantón Salcedo, ubicados al sur-oriente de la provincia de Cotopaxi, en la región interandina o Sierra del Ecuador. Particularmente, en la parroquia urbana San Miguel y las parroquias rurales: Antonio José Holguín, Cusubamba, Mulalillo, Mulliquindil y Panzaleo.
2.1 Muestreo
Fueron muestreados 106 caninos domésticos (con hogar y callejeros) de diferentes edades y sectores de la zona de estudio. Creo que aquí se podría indicar si los animales son domésticos o callejeros (de ser posible).
Para determinar la presencia de Toxocara canis en los caninos se utilizó la técnica de flotación de Sheather (método de concentración por flotación con centrifugación en una solución de azúcar), que permite diagnosticar diversas infecciones parasitarias en perros, gatos y otras especies.
Los caninos positivos al examen coprológico fueron desparasitados, se empleó Bongo Forte (Pamoato de pirantel 75 mg, Praziquantel 25 mg, aceite de hígado de bacalao 40 mg, equivalentes a 1 ml x cada 5 kg de peso.
La recolección de los parásitos adultos se realizó al siguiente día, se obtuvieron entre 2 a 6 parásitos de cada canino, expulsados en las deposiciones. Los parásitos fueron lavados para evitar que la materia fecal dañe la estructura y afecte las siguientes fases del procedimiento. Posteriormente, los parásitos fueron colocados en frascos con alcohol al 70%, debidamente rotulados con el código que identifica la zona y la edad del canino.
2.2 Disección de los parásitos adultos
La preparación de las muestras se realizó en el Laboratorio de Biología y Parasitología de la Universidad Técnica de Cotopaxi, fueron seleccionados parásitos adultos machos, diferenciados de las hembras por su morfología. Se colocó cada parásito en una placa de Petri, identificando la cola y diseccionando el último tercio. Con una balanza analítica se procedió a pesar la parte diseccionada, realizando varios cortes. Para el envío al laboratorio de las porciones seleccionadas, se las colocó en tubos Eppendorf (microtubo) identificados con sucódigo Tox-S, añadiendo 1 ml de alcohol al 70% para garantizar la conservación de las muestras hasta el análisis en el laboratorio de genética molecular.
2.3 Extracción de ADN
El análisis molecular de las muestras se realizó en el Laboratorio de Investigación de la Universidad de las Américas, UDLA (Quito, Ecuador).
Fue extraído el ADN de una porción de tejido de cada parásito mediante el método fenol-cloroformo:
Diseccionar 2 mm de la muestra de tejido y transferirla a un nuevo tubo de 1,5 mL.
Añadir 500 μL de buffer de extracción y macerar con una punta de pipeta o pistilo por 5 minutos al interior de un bloque frío.
Realizar mezcla (vortex) por 1 minuto y colocar 5 μL de proteinasa K (20 mg/mL).
Incubar las muestras a 56°C toda la noche en agitación, a 300 rpm.
Extraer las muestras del termobloque, dejar reposar 5 minutos a temperatura ambiente.
Añadir 750 μL de fenol/cloroformo/isoamílico y realizar vortex hasta formar una emulsión lechosa.
Centrifugar 10 minutos a 4°C a máxima velocidad. Transferir toda la fase acuosa a un nuevo microtubo de 1,5 mL.
Añadir a esa fase, 500 μL de cloroformo: isoamílico, homogeneizar en vortex.
Centrifugar 10 minutos a 4°C a máxima velocidad. Transferir toda la fase acuosa a un nuevo microtubo de 1,5 mL.
Añadir al nuevo microtubo, 400 μL isopropanol 100 % y 40 μL de NaCl 3M. Agitar bien y dejar reposar a temperatura ambiente por dos horas.
Centrifugar por 30 minutos a 4°C a 15000 rpm.
Descartar el sobrenadante con mucho cuidado de no alterar el pellet.
Agregar 1.000 uL de etanol 70 % helado y centrifugar por 15 min a 15000 rpm.
Descartar el sobrenadante y dejar secar.
Resuspender en 40 μL de agua Milli Q o TE e incubar la muestra a 37°C por dos horas.
Cuantificar la concentración de ADN.
2.4 Cuantificación y dilución de muestras de ADN
La cuantificación de las concentraciones de ADN se realizó mediante un espectrofotómetro (NanoDrop), posteriormente se procedió a diluir cada muestra hasta alcanzar una concentración final de 5 ng/uL(Tabla 1). Las muestras fueron almacenadas a -20oC hasta su uso.
2.5 Amplificación de los fragmentos
La amplificación por PCR de la región del espaciador transcrito interno (ITS-2) del ADN ribosómico (ADNr) se realizó utilizando el par de cebadores (primers) específicos para la especie parasitaria en estudio, estos fueron diseñados por investigadores de la Universidad Técnica de Cotopaxi-UTC y la Universidad de las Américas-UDLA:
Las reacciones de PCR fueron realizadas a un volumen final de 15 μl para cada muestra (Tabla 2).
Los fragmentos generados por PCR mediante el uso de termocicladores, siguieron las siguientes condiciones: temperatura inicial de desnaturalización de 94°C por 2 min (1 ciclo), seguido de 35 ciclos continuos de (desnaturalización a 94°C por 1 min, acoplamiento de los primers a 58°C por 30 seg, extensión a 72°C por 1 min y una extensión final de los productos a 72°C por 5 min), finalizando con10 min de reposo a 4°C.
Los amplicones fueron visualizados en geles de agarosa al 2%, para verificar su presencia. Los geles fueron coloreados con SYBR Safe, sobre luz ultravioleta.. En cada caso se corrió un control negativo para verificación de contaminación
2.6 Purificación y Secuenciación
El producto amplificado fue secuenciado con kit de secuenciación BigDye v3.1 (Applied Biosystems). Las reacciones de secuenciación fueron purificadas mediante acción enzimática, protocolo basado en resina de filtración en gel. Se empleó la matriz de secuenciación Sanger (BigDye - 3.1), desarrollando la secuenciación en electroforesis capilar.
2.7 Alineamiento de las secuencias
Las secuencias del gen ITS-2 (ADNr) de T. canis obtenidas en el estudio fueron editadas y alineadas inicialmente en el programa MEGA X [5], a partir de la secuencia de referencia (MK309928.1) disponible en el GenBank [6].
2.8 Análisis estadísticos
2.8.1 Identificación molecular
La identidad de cada uno de los aislados fue confirmada mediante una búsqueda de similitud, al comparar su secuencia de nucleótidos con otras secuencias de nematodos ascaridoides disponibles en el GenBank, a partir del algoritmo NCBI BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi).
2.8.2 Análisis Filogenético
Los árboles filogenéticos fueron generados entre secuencias de la región del espaciador transcrito interno (ITS-2) del ADN ribosómico (ADNr) de T. canis, pertenecientes al estudio y la comparación con otras secuencias depositadas en el GenBank con los números de acceso: MK447598, MK447599 [7], MK309928 [6], MK728991, MK728992 [8], JN617989 [9], KP406763 [10], para Toxocara canis; KY003089, KY003091, KY003065, KY003080 [11] para Toxocara cati; KT737382 [12], FJ418784 [13], KJ777160 [14], MK100346 [15] pertenecientes a Toxocara vitulorum y MT341323 [16] secuencia de Ascaris suum. La construcción de dichos árboles se desarrolló utilizando el método NJ (neighbor-joining). La confiabilidad de los árboles obtenidos se determinó con un test de replicaciones (bootstraping) con 1 000 réplicas, mostrado junto a las ramas. Todos los análisis se realizaron en el programa MEGA X [5].
3. Resultados y discusión
La población de caninos estudiados pertenecía a diferentes edades, desarrollados en variadas condiciones de vida lo que podría afectar el grado de parasitosis. Del total de perros estudiados (106 animales), 16 resultaron positivos a la presencia de huevos de Toxocara canis, representado un 16.96 % de prevalencia.
La distribución a nivel global de la infección por Toxocara canis en perros varía de 0 a 99,4 %, con altas tasas de prevalencia en perros y en humanos en Latinoamérica, tal como ha sido descrito por Guarín (2014) al realizar una revisión sistemática y evaluando la situación de la toxocariasis en algunos países de Latinoamérica. Particularmente en países como Argentina se han reportado valores de prevalencia que oscilan entre 9 y 19 %, Brasil en el rango de 5 - 39 % y Chile de 13,5 - 19 %, a partir de hallazgos reportados en estudios epidemiológicos de prevalencia de toxocariasis, encontrando parásitos adultos y huevos en muestras fecales de perros y huevos en suelos, en plazas públicas y parques de zonas urbanas, fundamentalmente. Evidenciando el problema de salud pública, que puede generarse por esta causa [17].
3.1 Identificación molecular
En el estudio fueron secuenciados un total de 383 pb de la región del gen ITS-2 del ADN ribosómico (ADNr) de T. canis aislados de caninos del cantón Salcedo, en Cotopaxi.
Los 20 aislados se identificaron como Toxocara canis cuando se compararon con secuencias previamente depositadas en el GenBank mediante el algoritmo BLAST (Figura 2), mostrando un 100 % de identidad para T. canis en relación a las secuencias MK728991 y MK728992, reportadas por Gnani [8] quien desarrolló la detección y caracterización molecular de helmintos de importancia zoonótica transmitidos por el suelo, en Andhra Pradesh, India.
Los resultados han servido como análisis primario para la confirmación de los aislamientos en caninos del cantón Salcedo como T. canis y pudiera constituirse como la primera caracterización molecular en adultos de T. canis en el Ecuador.
Siendo de mayor importancia, al considerar los criterios de Huang et al. (2017), quienes demuestran que las herramientas moleculares como la PCR convencional resultan de gran importancia en el diagnóstico de especies parasitarias como los nematodos, debido a su elevada especificidad y sensibilidad [18], muy útiles en casos donde las similitudes entre los huevos y los estados larvarios de los helmintos entorpecen la identificación entre especies.
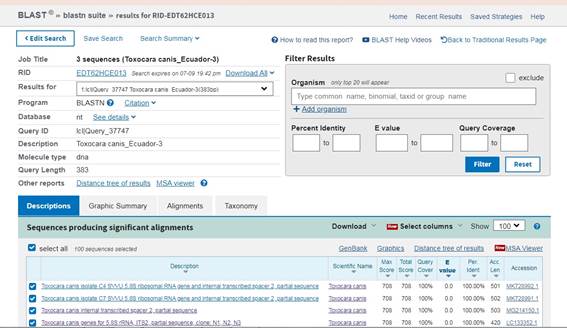
Fig. 1: BLAST (porcentaje de identidad) entre secuencias de ITS-2 del ADNr (383 pb) de los aislados Toxocaras canis y otras secuencias recuperadas del GenBank.
El presente estudio confirmó que la secuencia de la región del espaciador transcrito interno (ITS-2) del ADN ribosómico (383 pb) de T. canis, es una región muy conservada (Figura 2), tal como había sido planteado en otros estudios donde se describe que la región ITS contiene dos regiones no codificadoras variables que se encuentran dentro de las repeticiones altamente conservadas del ADNr, entre la subunidad pequeña, la región para 5.8S y los genes para ADNr de la subunidad grande [19].

Fig. 2: Alineamiento entre secuencias de ITS-2 del ADNr (383 pb) de los aislados Toxocara canis y otras secuencias recuperadas del GenBank.
Por ello, estas regiones del ADNr son las comúnmente estudiadas entre los tres genes codificantes (18S, 5.8S, y 28S) y los dos espaciadores internos (ITS1 e ITS2). Aspecto también descrito por Wickramasinghe et al., (2009) a partir de la caracterización molecular, discriminación y análisis filogenético del Toxocara canis y Toxocara vitulorum, basado en genes mitocondriales y ribosomales, entre ellos el ITS-2 y 28S [13].
Estudios basados en el ADN para identificar las especies de nematodos, particularmente utilizando fragmentos de la región ITS del ADN ribosomal demuestran la eficacia de las técnicas moleculares en estudios epidemiológicos [20]. Otros análisis genéticos desarrollados en la provincia de Guangdong en China, permitieron dilucidar la epidemiología, la genética y la biología del Toxocara cati [11]. Es por ello que, las secuencias altamente conservadas del gen 5.8S, han sido utilizadas como punto de unión de cebadores universales para amplificar las regiones espaciadoras flanqueantes, y que el ITS1 e ITS2 resultan de gran utilidad para establecer relaciones filogenéticas. Quedando demostrado la importancia de los estudios basados en el empleo de la reacción en cadena de la polimerasa, ya sea utilizando marcadores genéticos del ADN ribosómico y/o del ADN mitocondrial, por ser efectivos en la identificación y diferenciación de ascaridoides a nivel de especie [5].
Coincidiendo con Pérez (2009) sobre la importancia de la identificación de parásitos cuando se suman otras técnicas como las moleculares, ya que permiten investigar diferencias entre algunas especies crípticas de parásitos [21], siendo aplicadas por ejemplo para estudiar la variación intraespecífica entre las secuencias ITS de Toxocara canis, Toxocara cati y Toxascaris leonina de diferentes especies hospedantes en el suroeste de Polonia, amplificando las regiones del espaciador transcrito interno (ITS)-1 e ITS-2 del ADN ribosómico, identificando con eficacia aspectos asociados a la variabilidad intraespecífica [20].
3.2 Análisis Filogenético
En estudios sobre filogenética de especies parasitarias, autores como Pérez-Tris et al., (2007) y Hellgren et al., (2007) han referido la trascendencia de las técnicas moleculares como apoyo de la taxonomía tradicional, donde las especies morfológicas forman grupos de linajes recíprocamente monofiléticos: grupos cuyos miembros descienden todos de un ancestro común [21, 22].
De esta forma, al realizar análisis que incluyen simultáneamente varias fuentes de información como es el caso de caracteres morfológicos y la secuenciación de ADN nuclear o mitocondrial, se logran mayores resultados, pues implica mayor rigor en la prueba de corroboración de caracteres.
A decir de Villalobos-Cid (2019), la comparación de topologías de árboles (como están unidas las ramas o el patrón de ramificación) puede ser utilizada para explorar la historia evolutiva global de la especie, abriendo la posibilidad de una evaluación más amplia [23].
En este sentido, el árbol filogenético resultante de esta investigación y basado en secuencias ITS-2 (Figura 3) muestra la formación de dos agrupamientos principales, el primero de ellos para las secuencias de Toxocara canis de aislamientos en caninos del cantón Salcedo junto a las secuencias de referencia de T. canis reportadas por varios autores [6, 10], confirmando la identidad de los aislados estudiados como pertenecientes al género Toxocara, particularmente la especie T. canis.
Las secuencias de Toxocara cati [12] y de Toxocara vitulorum [13, 14] se muestran distantes formando grupos independientes, lo cual corrobora la identificación del T. canis. La secuencia de Ascaris suum [16] se emplea como taxón externo al grupo de estudio.

Fig. 3: Árbol de relaciones filogenéticas según el algoritmo neighbor-joinin, basado en las secuencias de ITS-2 del ADNr (383 pb) de los aislados Toxocaras canis y otras secuencias recuperadas del GenBank.
Los resultados obtenidos coinciden con los reportados por Mahdy et al., (2020) quien al realizar la caracterización molecular y el análisis filogenético de especies de Toxocara en perros, bovinos y búfalos en Egipto, concluyen que el gen ITS-2 es una buena herramienta sensible para la identificación de gusanos adultos de Toxocara spp., diferenciando al T. vitulorum del T. canis en su estudio [24]
De igual manera, Pawar et al., (2012) realizaron estudios del ADN amplificando la región del espaciador transcrito interno (ITS-2) del ADN ribosómico, una región génica más apropiada y válida para diferenciar entre especies de Toxocara (Toxascaris leonina y Toxocara cati) [25].
En relación a este criterio otros autores demostraron que el ADN ribosómico (ADNr) es un marcador valioso, empleado frecuentemente para conseguir la identificación de parásitos de especies en Toxocara y Toxascaris [4, 6].
El árbol de relaciones filogenéticas desarrollado por Mahdy et al., (2020), demuestra de forma clara el T. vitulorum y el T. canis forman dos agrupaciones, evidenciando la existencia de dos linajes diferentes [24].











 uBio
uBio