INTRODUCCIÓN
El consumo de hortalizas verduras es vital para la salud humana puesto que poseen innumerables propiedades alimenticias, son fuente inagotable de vitaminas, minerales, fibra y energía; y pueden contribuir de alguna forma con la prevención de enfermedades cardiovasculares y gastrointestinales 1. Sin embargo, por sus características físicas y de cultivo, estos productos están expuestos a contaminación de tipo biológica y química, constituyendo un riesgo para la adquisición de enfermedades transmitidas por alimentos (ETA) 2.
La contaminación de los productos hortofrutícolas ocurre a partir de diversas fuentes como: uso de agua de riego contaminada, el suelo de cultivo, la materia fecal humana o animal, el aire, el equipo de cultivo y manejo, los recipientes y utensilios, los materiales de transporte y el humano. El consumo de vegetales crudos ha sido asociado a numerosos casos de brotes de enfermedades por microorganismos patógenos como Listeria monocytogenes, Clostridium botulinum, Vibrio cholerae y Escherichia coli3.
Las ETA son consideradas un problema de salud pública a escala mundial, siendo el agua y los alimentos las principales fuentes de enfermedades entéricas agudas. Se han descrito alrededor de 200 enfermedades de transmisión alimentaria, cuya etiología incluye bacterias, virus, hongos, parásitos, productos químicos y toxinas de origen vegetal. Se estima que anualmente se enferman unos 600 millones de personas (1 de cada 10 habitantes) por ingerir alimentos contaminados. Los niños menores de 5 años cerca de un 40% contraen dicha enfermedad, que provocan cada año 125 000 defunciones 2-5.
Ecuador es un país potencialmente agrícola con productos de primera necesidad que además son de exportación a nivel mundial 6. Es por ello, que se implementó un plan para mitigar la aparición de bacterias multiresistentes, proyecto presentado por La Organización de las Naciones Unidas para la Alimentación y la Agricultura (FAO) con el propósito de brindar asistencia técnica a los gobiernos para formular e implementar planes nacionales multisectoriales para controlar la resistencia antimicrobiana en la alimentación y la agricultura. Dentro de las principales fuentes de contaminación en la agricultura se encuentra el uso de agua de riego ya que ésta pudiera ser no potable, además, la ubicación geográfica de las huertas debido a que estas pudieran estar cerca de aguas residuales, proviniendo quizá de hospitales, casas, restaurantes, escuelas. En el agua de riego se encuentran bacterias patógenas, las cuales al poseer genes de virulencia y de resistencia a antibióticos pudieran transferir estos genes a bacterias no patógenas también presentes en estos ambientes 5.
Tomando en cuenta que los vegetales frescos forman parte esencial de la dieta humana y además pueden estar asociados con la transmisión de bacterias multiresistentes, el objetivo de esta investigación fue identificar los perfiles de resis tencia antimicrobiana de bacterias aisladas en cultivos agrícolas de la cuenca del río Chambo.
MATERIALES Y MÉTODOS
Sitios de muestreo
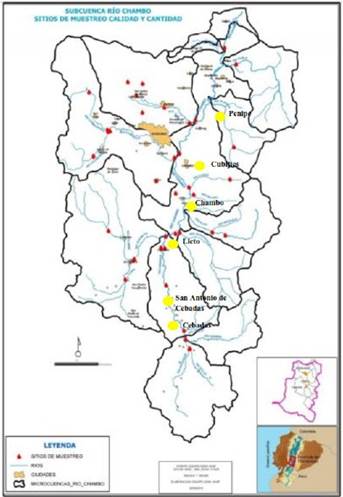
Se analizaron un total de 12 muestras de vegetales y hortalizas encontrados en las cercanías (500 m) de seis zonas agrícolas identificadas como puntos de recolección, cuyos productos son regados directamente del caudal del río Chambo, el cual cubre una superficie aproximada de 3.580 Km2. El recorrido de este río atraviesa la provincia de Chimborazo que consta de cantones como: Penipe, Guamote, Alausí, Chambo, Colta, Riobamba y Guano. Donde se fijaron los seis puntos para la recolec ción de los diferentes cultivos agrícolas (Figura 1).
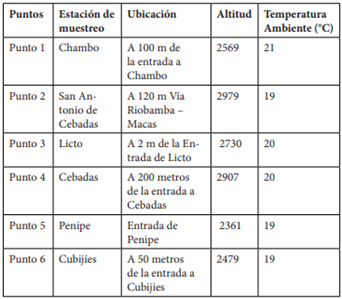
Se tomó en cuenta la altitud y la variante clima tológica temperatura de las zonas, este estudio se ejecutó durante el período abril - julio de 2019 (Tabla 1).
Muestras
Las 12 muestras de vegetales y hortalizas se tomaron por duplicado de manera aséptica en bolsas estériles, posteriormente se selló, se asignó su respectiva codificación a cada bolsa y se conservó en refrigeración en una cava con hielo hasta el momento de su llegada al laboratorio de Microbiología, Facultad de Ciencias de la Salud, en la Universidad Nacional de Chimborazo, establecimiento donde se llevó a cabo análisis microbiológico.
Cultivo y aislamiento de cepas bacterianas presentes en los vegetales y hortalizas
Una vez que las muestras de productos vegetales fueron transportadas al laboratorio de Microbiología, se realizó el análisis microbiológico. Del centro del fruto del producto agrícola se tomó 25 g (triturado) y se colocó en 225 ml de agua peptonada para realizar el preenriquecimiento y luego se incubaron por 24 horas a 37°C. Posteriormente se tomó 1ml de cultivo y se inoculó en un tubo con 9 ml de agua peptonada los cuales se incubaron 24 horas a 37°C. Transcurrido el periodo de incubación se tomaron 10 microlitos del cultivo para sembrar por la técnica de agotamiento en los medios de cultivo agar MacConkey (MK) Acumedia©, Agar Sangre (AS) Himedia©, Cistina Electrolito Deficiente (CLED) DifcoTM y Tiosulfato Citrato Bilis Sacarosa (TCBS) DifcoTM.
Preparación de medios de cultivo
Los medios de cultivo fueron preparados según instrucciones del fabricante. Todos los medios fueron autoclavados a 15 psi a 121 °C durante 25 minutos, se enfrió y se colocó en cajas monopetry Greiner© estériles, con un volumen aproximado de 15 mL sobre una superficie horizontal y en tubos anteriormente esterilizados un volumen de 5 ml respectivamente. Posteriormente solidificado el medio de cultivo se procedió almacenar en fundas de estériles para evitar la posible contaminación a una temperatura de 2 - 8°C.
Técnica de aislamiento de colonias
La técnica empleada fue la siembra por agotamiento en agar. Una vez obtenidas las colonias seguidamen te se realizó la tinción de Gram y pruebas bioquímicas para la identificación bacteriana a través de métodos microbiológicas convencionales de acuerdo con los esquemas de Koneman et al 6.
Pruebas bioquímicas para la identificación de bacterias:
Las pruebas bioquímicas utilizadas para las bacterias Gram positivas fueron la observación de α, β, o γ hemólisis en agar sangre, y las pruebas de catalasa, agar bilis esculina y crecimiento en caldo BHI con NaCl al 6,5%.6.
En el caso de las bacterias Gram negativas se realizaron las pruebas: oxidasa, agar kliger, agar urea, citrato, caldo malonato, agar motilidad-in dol-ornitina (MIO) y agar lisina hierro agar (LIA) 6. El agar Kligler se utilizó para observar la fermentación de la glucosa o la lactosa, la presencia de H2S y si había producción de gas o no. Se utilizó la urea para ver la producción de ureasa, el agar citrato como fuente de carbono y nitrógeno. El caldo Malonato se utilizó para diferenciar microorganismos entéricos según su capacidad de utilizar el manitol. Para observar la motilidad bacteriana, la producción de indol y la actividad enzimática ornitina descarboxilasa se usó el medio MIO y para detectar la desaminación o descarboxilación de la lisina se probó el medio LIA.
Determinación del perfil de susceptibilidad a los antibióticos de las cepas bacterianas aisladas e identificadas
A las bacterias aisladas, se les realizó ensayos de sensibilidad por el método de difusión del disco en agar Mueller Hinton (Oxoid Basingstoke, UK) de acuerdo a las instrucciones del Clinical and Laboratory Standard Institute (CLSI, 2019) 7.
Para realizar la prueba se transfirió una o dos colonias del cultivo a un tubo con solución fisiológica estéril, el crecimiento bacteriano se ajustó a la turbidez del patrón 0.5 del estándar de McFarland. Se introdujo un hisopo de algodón estéril dentro del tubo que contenía el inóculo estandarizado. Con el hisopo humedecido, se inoculó en tres o cuatro direcciones toda la superficie de una placa con agar Mueller Hinton. Se dejó secar el inóculo a temperatura ambiente durante 5 minutos. Posteriormente se procedió a la colocación de los discos de antibióticos con una pinza esté ril luego se incubaron las placas 24 horas a 37°C. Transcurrido el período de incubación se realizó la lectura midiendo con una regla milimetrada, la zona clara alrededor del disco de antibiótico, el cual se corresponde con la inhibición del crecimiento bacteriano. Estos datos se compararon con los diámetros de zona establecidos para cada antibiótico en las tablas de interpretación internacional del CLSI 2019. La interpretación de los halos de inhibición permitieron expresar los re sultados como sensible o resistente.
Se ensayaron los siguientes antimicrobianos (BD BBLTM, USA): ceftazidima (CAZ) 30 µg, Cef triaxona (CRO) 30 µg, trimetoprim-sulfa (SXT) x µg, penicilina (P) 10 U, imipenem (IMI) 10 µg, tetraciclina (TE) 30 µg, ciprofloxacina (CIP) 5 µg, gentamicina (GM) 10 µg, Amikacina (AK) 30 µg, vancomicina (Va) 10 µg.
Para estos ensayos se utilizó como cepa control P. aeruginosa ATCC 25923, S. aureus ATCC 25923, K. pneumoniae ATCC 700603.
RESULTADOS
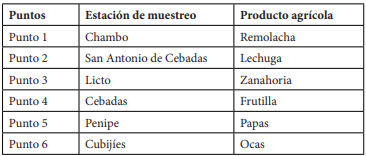
Se seleccionaron puntos estratégicos tomando como referencia la longitud del río y las zonas agrícolas de los distintos sectores para la recolec ción de productos cercanos al río Chambo. Los puntos fueron: Chambo, San Antonio de Cebadas, Licto, Cebadas, Penipe y Cubijíes (Tabla 2).
Se aislaron e identificaron de los productos agrícolas procesados un total de 14 bacterias; 12/14 (85,71%) pertenecientes a la familia Enterobacteriacea y 2/14 (14,28%) a la familia Enterococca ceae.
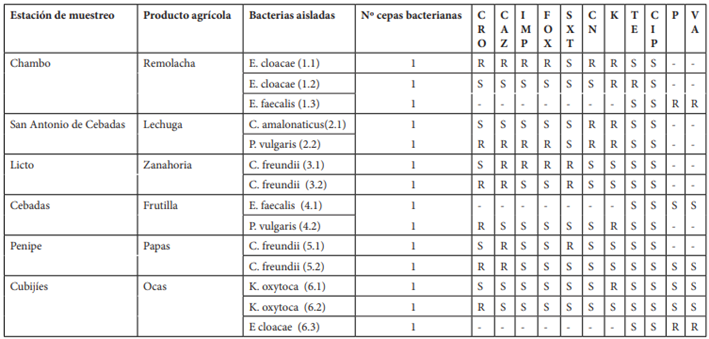
De la Remolacha se aislaron 2 cepas de Entero-bacter cloacae y 1 de Enterococcus faecalis. De la lechuga se aisló 1 cepa de Citobacter amalonaticus y 1 de Proteus vulgaris. De las zanahorias 2 cepas de Citrobacter freundii. De la frutilla, 1 cepa de E. faecalis y 1 de P. vulgaris. De las papas, 2 cepas de C. freundii y de las ocas, 2 cepas de Klebsiella oxytoca y 1 de E. cloacae (Tabla 3).
El 50% (6/12) de las enterobacterias aisladas de estos productos agrícolas presentaron resistencia a cefalosporinas de 3ra generación (ceftriaxona y ceftazidima), 50% (6/12) a kanamicina y 25% (3/12) a imipenem, gentamicina y trimetoprim sulfametoxazol (tabla 3).
Con respecto a la multirresistencia de las cepas de Enterobacterias estudiadas, el 16,7 % (2/12) resultaron resistentes a seis antibióticos, 8,3 % (1/12) a cuatro antibióticos, 8,3% (1/12) a tres antibióticos y el 41,7 % (5/12) a dos antibióticos (tabla 3).
Tabla 3 Perfiles de susceptibilidad a los antibióticos de cepas bacterianas aisladas de los productos agrícolas de la Cuenca del río Chambo.
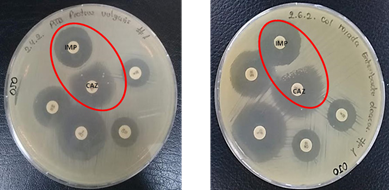
Tres cepas expresaron fenotipos correspondientes a la producción de betalactamasas AmpC inducibles. Este mecanismo AmpC tipo cromosómico inducible, se pudo evidenciar fenotípicamente utilizando como agente inductor al imipenem y como sustrato a ceftazidima observándose un achatamiento en forma de D en el halo de inhibición de ceftazidima (Figura 2).
DISCUSIÓN
Los alimentos vegetales son una de las principales vías de transmisión de microorganismos a través de la cadena alimentaria. En la superficie de las plantas en crecimiento existe una microbiota típica, que se puede contaminar por el aporte de microorganismos de distinta procedencia, como puede ser el origen animal que aporta al suelo y al agua, sus excretas 2.
Otra vía de contaminación son las aguas residuales y de riego, ya que los cultivos pueden ser regados por estas aguas sin tratar, existe la posibilidad de que los alimentos vegetales recién cosechados estén contaminados por microorganismos patógenos para el hombre, sobre todo por aquellos que producen trastornos gastrointestinales 8.
La subcuenca del Río Chambo se encuentra ubicada en el centro del Ecuador, teniendo una superficie de 3580 km2 y cubre un 54% de la superficie total de la provincia de Chimborazo, en esta zona se encuentra parte de los siete cantones de la provincia: Alausí, Riobamba, Chambo, Penipe, Guano, Colta y Guamote, donde se encuentran unos 33 ríos de tamaño variable según su ubicación geográfica y época del año. La subcuenca pertenece a la provincia en un 98% abarcando los humedales y zonas naturales, como los páramos (9.
En esta subcuenca se han desarrollado algunos estudios para conocer la calidad del agua en toda su extensión. El equipo técnico de la Central Ecuatoriana de Servicios Agrícolas-Agrónomos y Veterinarios Sin Fronteras (CESAA- VSF) realizaron por primera vez estudios en 33 estaciones seleccionadas con dos muestres cada una. Como primeros indicadores de la calidad del agua se analizaron diversos parámetros tanto físicos, químicos y microbiológicos, evaluándose el oxígeno disuelto, pH, temperatura, PO4, NO3, turbidez, conductividad, salinidad, solidos totales disueltos, presencia de coliformes totales y E. coli como principal indicador microbiológico de contaminación. Los resultados obtenidos de los distintos parámetros obtenidos confirmaron que los ríos Guano, Guate, Chibunga y Chambo se encuentran contaminados 10,11.
En esta subcuenca donde se ubican los sitios agrícolas de muestreo no han sido desarrollados ningún estudio para evaluar los productos cosechados. Sin embargo, se ha reportado un trabajo similar donde se identificaron bacterias de interés clínico como: C. diversus, C. amalonaticus, E. aerogenes, E. cloacae, Hafnia alvei, Aeromona spp y Enterococcus spp aisladas de productos agrícolas regados con aguas del río Guano, Chimbora zo, Ecuador 12.
En las zonas agrícolas seleccionadas (Chambo, San Antonio de Cebadas, Licto, Cebadas, Penipe y Cubijíes) cercanas al río Chambo donde se recolectaron los productos se siembran vegetales y hortalizas que se expenden a los mercados de Riobamba. Los agricultores de estas zonas implementan prácticas de producción orgánica. Los pequeños productores de estas zonas hacen un manejo racional de los recursos naturales, sin la utilización de productos de síntesis química para la obtención de alimentos sanos. Se ha descrito que en diversos puntos del río Chambo existe la presencia de coliformes fecales y E. coli como bacteria indicadoras de contaminación fecal, esto debido tanto a actividades humanas y ganaderas que se encuentren próximas al río, los productos agrícolas de estos sectores son regados con estas aguas contaminadas de donde se asume que llegan las bacterias multiresistentes 13.
Las bacterias más prevalentes descritas tanto en estudios realizados en el agua como en los productos agrícolas regados con las aguas de los ríos Chambo y Guano son enterobacterias y E. faeca lis (11,12.
En el presente estudio, se encontró que el 83,3% de las bacterias aisladas de los productos agrícolas obtenidos de los puntos cercanos al río Chambo correspondían a enterobacterias de los siguientes géneros: Citrobacter, Enterobacter, Klebsiella y Proteus bacterias comúnmente encontradas en agua, suelos, vegetación y microbiota intestinal de animales 14. Considerados potenciales bioindicadores de calidad del agua con bajo potencial patógeno y corresponden a 10% de los microorganismos intestinales humanos y anima les, por lo que su presencia en el agua está asociada con contaminación fecal e indica tratamientos inadecuados o contaminación posterior 15.
Por otro lado, en los productos como las remolachas y ocas se aisló cepas de E. faecalis la presencia de esta bacteria es considerado como un indicador de contaminación fecal de fuentes humanas 16. También está involucrada en el deterioro de los alimentos, en intoxicaciones alimentarias y en la propagación de resistencia a los antibióticos a través de la cadena alimentaria (17.
La presencia de Enterococcus spp. en ambientes tales como el agua, alimentos y el aire pudiesen estar relacionados a su transmisión, en especial con diseminación tanto de patrones de resistencia a antibióticos como en la producción de enfermedades 17.
El uso de antibióticos en diversas áreas incluyendo la agrícola ha aumentado lo cual pudiera facilitar la adquisición de resistencia a los mismos por parte de bacterias presentes en dicho ambiente, sean patógenas o no. Dicha adquisición de resistencia pudiera realizarse a través de la transferencia horizontal de elementos genéticos móviles tales como transposones, plásmidos o integrones 18,19.
El uso inadecuado de antibióticos en medicina humana, veterinaria y agrícola propicia la liberación de estos antibióticos al medio ambiente. Junto con estos antibióticos, bacterias con resistencia a ellos se pudieran liberar en las aguas que pudieran irrigar diversos cultivos 20.
Se cree que las plantas de tratamiento de aguas residuales son probables puntos críticos para la diseminación de la resistencia a los antibióticos en el medio ambiente, ya que ofrecen condiciones convenientes para la proliferación de resistencia bacteriana, así como para la transferencia horizontal de genes de resistencia entre diferentes microorganismos. De hecho, genes que confieren resistencia a todas las clases de antibióticos junto con elementos genéticos móviles como plásmidos, transposones, bacteriófagos, integrones se detectan en bacterias aisladas de las plantas de tratamiento de aguas residuales de diferentes países. Parece que estas plantas con procesos de tratamiento convencionales son capaces de reducir significativamente la resistencia bacteriana a los antibióticos pero no son eficientes en la eliminación de genes de resistencia. La implementación de procesos avanzados de limpieza de aguas residuales además de un tratamiento de aguas residuales convencional es un paso importante para proteger el medio ambiente acuático 20.
Estudios previos con respecto a la incidencia de bacterias con resistencia a antibióticos provenientes de agua de riego y productos agrícolas incluyen la búsqueda de resistencia a antibióticos en diferentes especies de Enterobacteriales y Enterococcus. En un estudio llevado a cabo por Abriouel et al., 21 donde analizaron diferentes cepas de enterobacterias y Enterococcus spp. provenientes de frutas, vegetales, agua y suelo reportaron resistencia a antibióticos como quinupristina/dalfopristina, estreptomicina, nitrofurantoína, levofloxacina, ciprofloxacina, rifampicina, cloranfenicol, tetraciclina, eritromicina y penicilina. En otro estudio realizado por Odonkor y Addo, 22 buscaron la prevalencia de resistencia a antibióticos en cepas de E. coli provenientes de fuentes de agua de riego, en las cua les, encontraron que 32.99% presentaba resistencia a penicilina, así como cefuroxima (28.87%), eritromicina (23.71%), tetraciclina (21.45%), ampicilina (11.32%) y ciprofloxacina (8.25%).
En el presente estudio, las Enterobacterias aisladas de los productos agrícolas presentaron resistencia a ceftriaxona (50%), ceftazidima (50%), kanamicina (50%), imipenem (25%), gentamicina (25%) y trimetoprim sulfametoxazol (25%). Es importante destacar que tres cepas de E. cloa cae (cepa 1.1), C. freundii (cepa 1.1) y P. vulgaris (cepa 2.2) expresaron fenotipos correspondientes a la producción de betalactamasas AmpC inducibles.
Las bacterias de los géneros Enterobacter y Citrobacter poseen de manera natural estas betalactamasas lo que explica su resistencia natural a las aminopenicilinas, cefalosporinas de 1ra genera ción, cefamicinas (cefoxitina, cefotetán) y aminopenicilinas combinadas con inhibidores de betalactamasas (amoxicilina-ácido clavulánico, ampicilina-sulbactam).
La cepa de P. vulgaris presentó fenotipo de resistencia presuntivo a betalactamasa tipo AmpC plasmidica inducible lo cual tendría que ser comprobado por métodos moleculares. Los genes ampC mediados por plásmidos han sido encontrados en bacterias como Salmonella spp., K. pneumoniae y P. mirabilis que naturalmente no poseen estos genes 23. La evidencia molecular sugiere que los genes que codifican a estas enzimas, derivan de los genes ampC cromosómicos que naturalmente poseen las enterobacterias de los géneros Enterobacter spp., Providencia spp., Morganella morganii, Serratia marcescens, C. fre undii y H. alvei Estos genes han sido integrados en elementos genéticos transferibles facilitando la diseminación a diferentes bacterias 24.
En relación a las 2 cepas de E. faecalis aisladas, presentaron resistencia a penicilina y vancomicina. La resistencia de Enterococcus spp. a diversos agentes antimicrobianos supone un problema de salud pública que afecta a todo el mundo, sobre todo a los países en los que el uso de antibióticos no está especialmente regulado 25.
La resistencia adquirida de Enterococcus a múlti ples antibióticos se ve favorecida por la acumulación de mutaciones y resistencias adicionales adquiridas a través de genes exógenos 16. El genoma de estas bacterias se caracteriza por poseer la capacidad de incorporar a través de elementos genéticos móviles exógenos, genes de resistencia a multitud de antibióticos, así como de transferir determinantes de resistencia generados por ellos mismos tras la exposición a ciertos antibióticos a otras especies patógenas 25,26.
La presencia de Enterococcus spp. en los alimentos también puede suponer una amenaza debido al impacto negativo que puede causar la dispersión de la resistencia a antibióticos a través de la cadena alimentaria. En alimentación animal se utilizan multitud de agentes antimicrobianos como terapia y profilaxis frente a infecciones bacterianas y para fomentar el crecimiento animal. Este uso excesivo de antibióticos en producción animal se relaciona directamente con el aumento de las resistencias a los antibióticos, encontrando similitudes entre las cepas de Enterococcus spp. de origen animal con cepas de Enterococcus spp. proveniente de infecciones humanas nosocomiales 27. Enterococcus spp., exhibe una variedad de mecanismos de resistencias intrínsecas y adquiridas frente a las mayores clases de antibióticos de uso clínico y están dotados de mecanismos de intercambio genético eficientes que facilitan la diseminación de los genes de resistencia a antibióticos 28. Las resistencias adquiridas especialmente frente a penicilina/am picilina, aminoglicósidos y glicopéptidos han ido en aumento, limitando su espectro terapéutico.
Existen evidencias científicas que relacionan el uso de antibióticos en la cría de animales con la aparición de cepas de Enterococcus spp., resistentes en los alimentos, corroborando la implicación de éstos en la diseminación de resistencias con repercusión en Salud Pública 29-30.
CONCLUSIONES
Los resultados de este estudio reflejan que existe contaminación de los cultivos agrícolas vertidos con agua de regadío de la cuenca del río Chambo por especies de enterobacterias y E. faecalis; con altos porcentajes de resistencia a antibióticos de uso clínico betalactamicos, aminoglucósidos y trimetoprim sulfa, además tres cepas de enterobacterias expresaron fenotipos de resistencia correspondientes a la producción de betalactamasas AmpC inducibles y 2 cepas de E. faecalis presentaron resistencia a vancomicina y penicilina, de allí la necesidad de considerar establecer criterios que requieren el esfuerzo conjunto de gobiernos, productores y consumidores, implementando procesos de tratamiento de aguas residuales eficientes, así como realizar un seguimiento microbiológico permanente teniendo en cuenta el aspecto de la resistencia a los antibióticos, junto con un mayor control de la ingesta de medicamentos y una adecuada gestión de los residuos médicos.