Forma sugerida de citar:
De la Torre Duque, D. I., Mafla Quezada, E. C., Puga Torres, B. H., Piantino Ferreira,A. J. 2018. Caracterización molecular del adenovirus aviar en pollos comerciales del Ecuador. La Granja: Revista de Ciencias de la Vida. Vol. 28 (2):84-91. http://doi.org/10.17163/lgr.n28.2018.06.
1 Introducción
El adenovirus aviar (FAdV) es un virus clasificado dentro del género Adenovirus en la familia Adenoviridae (King et al., 2012). Este género posee tres grupos clasificados como: FAdV-I (causante de HPS: Síndrome del Hidropericardio e IBH: Hepatitis por Cuerpos de Inclusión), FAdV-II (causante de HEV: Enteritis Hemorrágica del pavo) y FAdV-III (causantede EDS: Síndrome de Baja Postura) (Hess, 2000). El FAdV-I ha sido clasificado en 5 especies (AE) y 12 serotipos (1–8a, 8b–11) (Adams et al., 2017).La partícula viral tiene forma icosaédrica, carece de cápsula y tiene un diámetro de 70 a 90 nm. El genoma viral está formado por una cadena doble de ADN con un tamaño de 45 kb aproximadamente (Jiang et al., 1999). El diseño de la cápsula viral consiste en la unión de 252 capsómeros, siendo 240 formados por la proteína Hexon y los 12 restantes por la proteína Penton. De cada vértice en donde se encuentrala proteína Penton, se desprenden dos fibras de diferente tamaño, las cuales están formadas de proteínas (proteínas de la fibra) que le otorgan la capacidad antigénica al virus (Adair y Fitzgerald, 2008).
El FAdV-I, ha sido asociado a dos principales enfermedades presentes en las aves comerciales, la Hepatitis por Cuerpos de Inclusión (IBH) que fue descrita por primera vez en el año de 1963 en Estados Unidos (Helmboldt y Frazier, 1963), y actualmente asociada a los 12 serotipos del FAdV-I, y el Síndrome del Hidropericardio (HPS) que es una enfermedad que afecta principalmente a pollos de engorde jóvenes de entre 3 y 6 semanas de edad, y que ha sido asociado al serotipo 4 del FAdV-I (Balamurugany Kataria, 2004). En la actualidad el virus se encuentra distribuido alrededor del mundo (Alemneshet al., 2012), siendo responsable por producir disminución en el consumo de alimento, aumento en la conversión alimenticia y de la mortalidad, y aunque existe la asociación de la IBH e HPS con enfermedades inmunodepresoras como la Enfermedad Infecciosa de la Bursa (IBV) y la Anemia Infecciosa de las Aves (CAV), se ha detectado al FAdV-1 como agente primario en el desarrollo de IBH e HPS sin la presencia de otras enfermedades concomitantes (Sentíes-Cué et al., 2010). Su forma de contagio es principalmente por vía oral, aunque la transmisión vertical es una forma importante de contagio debido a un sistema inmunológico poco desarrollado de la progenie (Toro et al., 2001; Nakamura et al., 2011).
Los brotes de Hepatitis por cuerpos de inclusión han sido reportados en varios países de América Latina como Brasil, Chile, Perú, Ecuador y México (Hess, 2000; Toro et al., 2001). Mazaheri et al. (1998) reportaron la presencia del serotipo 4 del FAdV-I en el Ecuador, al ser aislado de tejidos hepáticos en aves con HPS y caracterizado por pruebas de neutralización y análisis por enzimas de restricción en el ADN viral (Toro et al., 2000). Dada la distribución global de todos los serotipos de este virus es importante conocer la distribución geográfica de cada serotipo dentro de nuestro país, por lo que el objetivo de este estudio se centra en la caracterización molecular de los FAdV-I que circulan actualmente en las granjas de aves comerciales del Ecuador, para contar con el conocimiento científico que permita tomar decisiones y aplicar medidas sanitarias en beneficiode la industria avícola de la región.
2 Metodologia
2.1 Muestras virales
Durante el año 2016 se colectaron muestras biológicas de diferentes granjas en la zona norte de la sierra ecuatoriana, cuyas aves presentaban sintomatología relacionada a hepatitis, mala absorción, aumento de la conversión alimenticia y aumento de la mortalidad. Las muestras colectadas fueron tomadas mediante la impronta en 13 tarjetas Whatman FTA (GE Healthcare Company, Little Chalfont, Buckinghamshire, UK) de órganos como riñones, tráqueas, bolsa de Fabricio, pulmones y tonsilas cecales de aves entre 4 y 7 semanas de edad, para luego ser transportadas hacia el laboratorio de Ornitopatología, en la Facultad de Medicina Veterinaria y Zootecnia de la Universidad de São Paulo en Brasil, en donde fueron realizados los análisis moleculares respectivos. Cada tarjeta FTA estaba impregnada con 2 a 4 muestras de diferentes órganos y diferentes aves, que correspondían a 13 granjas distintas.
2.2 Maceración y extracción viral
El material fue procesado dentro de un flujo laminar, desinfectado con alcohol a 70° y exposición directa a luz ultravioleta (UVc) durante 15 minutos. Se recortó todo del material impregnado en cada tarjeta FTA para la obtención final de 13 muestras diferentes que se colocaron en microtubos de 2 ml con PBS (Phosphate Bufered Saline), 0,1 M, pH 7,4 enproporción 1:1. La maceración se realizó con la utilización del Tissuelyser LT Bead Mill (Qiagen, Hilden,Germany) a 50 oscilaciones por segundo durante 5 minutos. El producto macerado fue centrifugado por 30 minutos a 12.000 x g y a 4C (Kooet al., 2013). Finalmente se colectó 200 L del sobrenadante para el aislamiento del material genético.
2.3 Extracción del ADN y Reacción en Ca- dena de la Polimerasa (PCR)
Para la extracción del material genético con fenol y cloroformo se utilizó la metodología descrita por Chomczynski (1993). Para la reacción de PCR fueron utilizados iniciadores diseñados por Meulemans et al. (2001), los cuales cubren parte de las regiones conservadas denominadas Pedestal 1 (P1) adyacentes a la región variable denominada Loop 1 (L1) en la secuencia de nucleótidos del gen Hexon, permitiendo amplificar los 12 diferentes serotipos del FAdV-I.
El mix de 25 L para la Reacción en Cadena de la Polimerasa (PCR) consistió de 1X de PCR buffer libre de Magnesio, 1,25 mM de cada desoxiribonucleótido trifosfato, 0,5 mM de cada iniciador, 1,25 Ude Platinum Taq polymerase (Invitrogen
Taq polymerase (Invitrogen by LifeTechnologies, Carlsbad, CA), y 2,5 L de ADN extraído a una concentración aproximada de 1.000ng/L. Las condiciones de amplificación fueron las siguientes: 94°C por 5 minutos, seguido de 35 ciclos de 95°C por 1 minuto, 52°C por 45 segundos y 72°C por 1 minuto, finalizando con una extensión final de 72°C por 10 minutos. El producto final fue sometido a electroforesis utilizando gel de agarosa a una concentración de 1,5% para verificar la amplificación de fragmentos de ADN con un tamaño de aproximadamente 897 pb.
by LifeTechnologies, Carlsbad, CA), y 2,5 L de ADN extraído a una concentración aproximada de 1.000ng/L. Las condiciones de amplificación fueron las siguientes: 94°C por 5 minutos, seguido de 35 ciclos de 95°C por 1 minuto, 52°C por 45 segundos y 72°C por 1 minuto, finalizando con una extensión final de 72°C por 10 minutos. El producto final fue sometido a electroforesis utilizando gel de agarosa a una concentración de 1,5% para verificar la amplificación de fragmentos de ADN con un tamaño de aproximadamente 897 pb.
2.4 Purificación y secuenciamiento
El producto amplificado fue purificado utilizando el GPXTM PCR DNA and Gel Band Purification kit (GE Healthcare, Piscataway, New Jerser, USA) según las instrucciones del fabricante. Cada producto purificado fue secuenciado en sentido senso y antisenso usando el BigDye Terminator Cycle Sequencing Kit v3.1 (Applied Biosystems por Life Technologies, Carlsbad, California, EUA). Las reacciones finales fueron analizadas en un ABI 3730 DNA Analyzer (Applied Biosystems por Life Technologies, Carlsbad, Califórnia, EUA).
Terminator Cycle Sequencing Kit v3.1 (Applied Biosystems por Life Technologies, Carlsbad, California, EUA). Las reacciones finales fueron analizadas en un ABI 3730 DNA Analyzer (Applied Biosystems por Life Technologies, Carlsbad, Califórnia, EUA).
2.5 Análisis filogenético
Las secuencias de nucleótidos fueron editadas usando el software CLC Main Workbench 7, y alineadas con el método CLUSTAL W disponible en el software Clustal X 2.0. El árbol filogenético fue inferido utilizando el método Maximum Likelihood basado en el modelo Tamura 3-parameter (Tamura, 1992), con1000 repeticiones, disponible en el software Mega 7. Para esto fueron usadas las secuencias de nucleótidos generadas en este estudio y secuencias internacionales de los serotipos 6 y 11 del FAdV-I, tomadas del GeneBank (National Center for Biotechnology Information). Las secuencias de referencia para el alineamiento y construcción del árbol filogenético fueron tomadas de Brasil, México, Estados Unidos,Canadá, Reino Unido, Europa, Bélgica, China,Japón, Korea del Sur y la India, cuyos números de acceso se encuentran descritos en la Figura 1.
3 Resultados y Discusión
3.1 Reacción en Cadena de la Polimerasa (PCR)
El producto final de la PCR para las 13 muestras fue sometido a electroforesis en gel de agarosa al 1,5%, con un marcador de peso molecular de 100 pb (100 bp DNA Ladder, Invitrogen TM), encontrando 4/13 resultados positivos (30,8%), al mostrar las bandas a la altura aproximada de 897 pb, las cuales fueron analizadas para la caracterización del respectivo serotipo (Mettifogo et al., 2014; Joubert et al., 2014; De la Torre et al., 2018). De acuerdo con Meulemans et al. (2001), el ADN amplificado mediante esta reacción puede incluir a los 12 serotipos de FAdV-I, sin poder determinar directamente el serotipo específico, a no ser que se utilicen enzimas de restricción para la obtención de bandas de ADN en gel de agarosa con tamaños específicos para cada serotipo (Figura 2) (Meulemans et al., 2001), o mediante el análisis de las secuencias de nucleótidos cuando son comparados con secuencias almacenadas en la base de datos del GeneBank. Mediante unanálisis virtual sobre el uso de enzimas de restricción,se pudo determinar que el uso de la enzima PstI, provocó un corte del ADN en el sitio de reconocimiento (CTGCAG) de las secuencias de 808 pbde la cepa CR119 de Japón, Bélgica y USP-EC-01,dando como resultado dos bandas de 786 pb y 26pb (Figura 2), a excepción de la cepa CR119 de Europa,que, por contar únicamente con 596 pb, no demostró regiones disponibles para la acción de cualquier enzima de restricción. Una enzima adicional EcoRI, realizó cortes del ADN en el sitio de reconocimiento (GAATTC) de las demás secuencias caracterizadas como serotipos 11 del FAdV-I, incluyendo la secuencia USP-EC-02, dando como resultado dos bandas de 746 bp y 63bp (Figura 2).
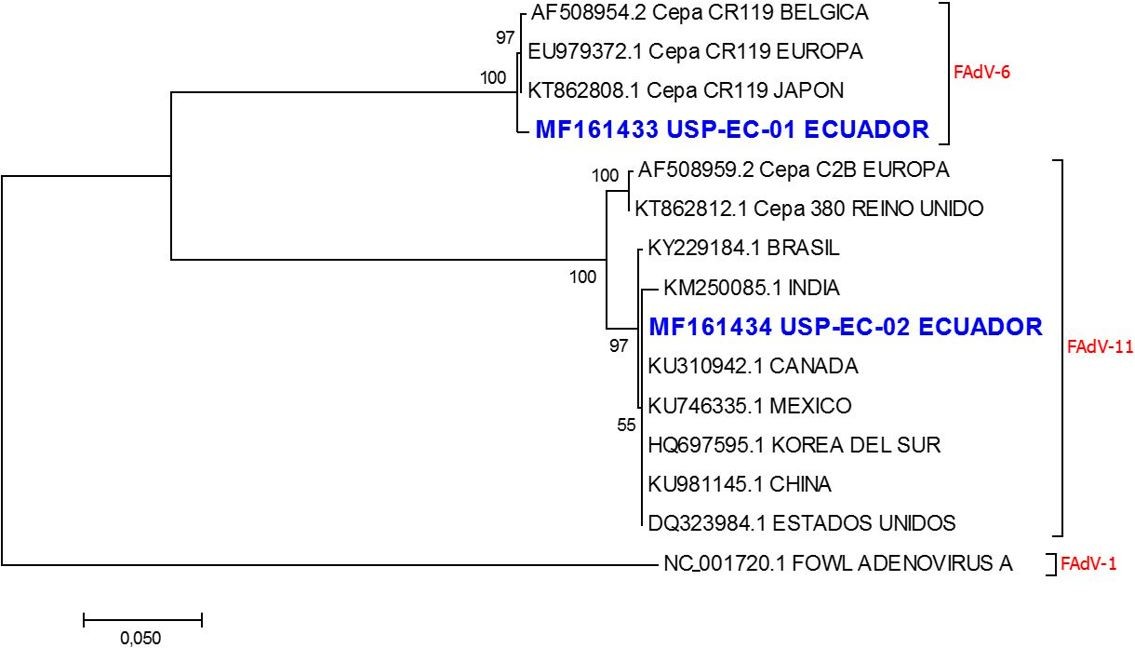
Figura 2. Árbol filogenético inferido usando el método Maximum Likelihood, basado en el modelo Tamura 3-parameter (Tamura, 1992). Las secuencias de las muestras de Ecuador están representadas en color azul y contienen su respectivo código de acceso en la base de datos del GeneBank. (FAdV=Adenovirus Aviar). La secuencia NC_001720.1 fue colocada como grupo externo.
3.2 Secuenciamiento
El secuenciamiento de los 4 productos positivos de la PCR dio lugar a la obtención de 2 diferentes secuencias y, siendo que tres muestras tuvieron secuencias exactamente iguales, se utilizó un consenso denominándolo USP-EC-02, y la secuencia de la cuarta muestra fue denominada USP-EC-01. Las secuencias obtenidas fueron ingresadas en la base de datos del GenBank con los códigos de acceso MF161433 para la secuencia USP-EC-01, y MF161434 para la secuencia USP-EC-02. De acuerdo a la matriz de identidad, las secuencias de nucleótidos (NT) y aminoácidos (AA) de USP-EC-01, tienen un alto porcentaje de identidad con las secuencias de las cepas CR119 del serotipo 6 del FAdV-I de Bélgica (99,1% de NT y AA), Europa y Japón (99,3% de NT y AA). Las secuencias de NT y AA de USPEC-02 tienen un alto porcentaje de identidad con las secuencias del serotipo 11 del FAdV-I de México, Estados Unidos, Canadá, Korea del Sur y China(100% de NT y AA), Brasil (99% de NT y AA), India (98,3% de NT y AA), Reino Unido (97,5% de NT yAA), y Europa (97,3% de NT y AA).
3.3 Análisis filogenético
Los resultados del análisis filogenético se muestran en la Figura 2. Todas las secuencias analizadas fueron agrupadas en dos grupos mayores que corresponden a los serotipos 6 y 11 del FAdV-I. La secuencia USP-EC-01, fue agrupada junto la cepa CR119 perteneciente al serotipo 6 del FAdV-I, registradas como originarias de Bélgica, Europa y Japón, demostrando la presencia de esta cepa viral en al menos los 3 continentes (Europa, Asia y América). La secuencia USP-EC-02, fue agrupada junto con secuencias de Europa, Reino Unido, Brasil, India, Canadá, México, Korea del Sur, China y Estados Uni- dos, las cuales han sido caracterizadas con el serotipo 11 del FAdV-I.
El FAdV-1 es responsable por causar dos principales enfermedades, la Hepatitis por Cuerpos de Inclusión (IBH) y el Síndrome del Hidropericardio (HPS) (Zhao et al., 2015). Hay algunos casos en donde el virus ha sido aislado de aves sin signos de enfermedad, por lo que se requiere un mayor desarrollo de técnicas que permitan diferenciar cepas patogénicas y no patogénicas dentro del mismo serotipo del virus (Absalón et al., 2017). En el Ecuador ya se ha reportado la presencia del serotipo 4 del FAdV- I como agente causal del Síndrome del Hidropericardio (Mazaheri et al.,1998), pero también es importante recalcar que todos los serotipos están relacionados con la IBH (Adair y Fitzgerald, 2008), y de esta forma se debe mantener un control epidemiológico de todas las cepas y serotipos que circulan por la región. 0 El serotipo 6 del FAdV-I pertenece a la especie E junto con los serotipos 7, 8a y 8b, pudiendo tener algún grado de protección cruzada con las vacunas contra el FAdV-I existentes en el Ecuador, que utilizan virus inactivados del serotipo 8, aunque esto no ha sido demostrado en ningún estudio experimental hasta el momento. Los serotipos 8b y11 demostraron provocar lesiones en hígado a nivel macro y microscópico, luego de ser inoculados experimentalmente por vía ocular, y sin la presencia de virus inmunodepresores (Steer et al., 2015), destacando que no existen desarrolladas vacunas específicas para el serotipo 11 que sean comercializadas en el Ecuador. Con estos resultados es importante considerar la necesidad del aislamiento y estudio experimental de los serotipos encontrados en el Ecuador con el objetivo de comprender la dinámica epidemiológica y la patogenicidad de todas las cepas de FAdV-I circulantes en el país.
4 Conclusiones
En este estudio se demostró la presencia de los serotipos 6 y 11 del Adenovirus Aviar, en lotes de aves comerciales del Ecuador. De esta forma se brinda un aporte a la comunidad científica para el desarrollo de nuevos estudios que permitan conocer la fisiopatología de las cepas circulantes y de ser necesario la creación de vacunas que ayuden a controlar la propagación y/o efecto negativo de estos virus sobre la producción avícola en las diferentes regiones del país.