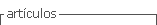INTRODUCCIÓN
El cáncer de pulmón es una de las neoplasias malignas con mayor incidencia en el mundo. En el año 2020 el Observatorio Mundial del Cáncer reportó que existe 2200000 nuevos casos y 1 800 000 muertes que ocasiona esta patología multifactorial 1. En América Latina, el cáncer de pulmón ha ocasionado 90550 muertes en el 2022, mientras que en nuestro país la neoplasia de pulmón refleja 1145 decesos 2. El desafío predominante asociado a esta patología radica en su notable incidencia y tasa de mortalidad en naciones de alto desarrollo socioeconómico. Es esencial priorizar la detección temprana, a pesar de que el Carcinoma de Pulmón de Células No Pequeñas suele diagnosticarse en etapas avanzadas de la enfermedad. La tos se erige como el síntoma más común, afectando entre el 50% y el 75% de los pacientes, seguido por la hemoptisis, el dolor torácico y la disnea 3.
El estudio de la enfermedad del cáncer de pulmón de células no pequeñas (NSCLC) representa una problemática de salud global de considerable magnitud, dada su prevalencia como el tipo más común de cáncer de pulmón y su complejidad clínica. Analizar detalladamente esta enfermedad es crucial por diversas razones 4. En primer lugar, la detección temprana del NSCLC presenta desafíos significativos, ya que los síntomas suelen manifestarse en etapas avanzadas de la enfermedad, lo que limita considerablemente las opciones de tratamiento. Los novedosos métodos de detección requieren mejorar, dado que se requiere un tratamiento personalizado en la terapia requerida. El hallazgo de nuevas dianas terapéuticas es importante debido a que la resistencia a las inmunoterapias tradicionales actualmente se consideran un obstáculo significativo.
El impacto socioeconómico del NSCLC es considerable, afectando no solo a los pacientes sino también a los sistemas de salud. Por lo tanto, analizar el NSCLC es esencial para mejorar las estrategias de prevención, diagnóstico y tratamiento, con el objetivo final de aumentar la vitalidad en los pacientes 4.
El cáncer de pulmón se clasifica en cáncer de pulmón de células no pequeñas (NSCLC) y en cáncer de pulmón de células pequeñas (SCLC) 5,6. El NSCLS abarca el 80% de los canceres de pulmón y se subdivide en adenocarcinoma, carcinoma de células grandes y carcinoma de células escamosas 6, mientras que el SCLC representa alrededor del 15% al 20% de todas las neoplasias pulmonares 5,6 y son considerados tumores de pulmón muy agresivos ocasionados por el hábito de fumar 5.
Los factores de riesgo en el cáncer de pulmón son el tabaquismo y la contaminación ambiental; a pesar de ello, distintos estudios han evidenciado que factores genéticos tienen un rol importante en el desarrollo de neoplasias de pulmón. Los factores ambientales y los eventos somáticos contribuyen al desarrollo de cáncer de pulmón esporádico. Aunque el hábito de fumar cigarrillos se considera un factor de riesgo primario, la incidencia de tumores de pulmón se ha elevado en personas que nunca han fumado 7.
Las alteraciones genéticas en el cáncer de pulmón radican en el receptor del factor de crecimiento epidérmico (EGFR). EGFR es una proteína tirosina quinasa receptora transmembrana que se expresa en tejidos epiteliales y mesenquimales normales. La sobreexpresión de esta proteína está asociada a la carcinogénesis 7. Las mutaciones del dominio quinasa de EGFR ocurren en los exones 18-21, lo que dirige a la hiperactivación de las vías de señalización pro-supervivencia posteriores dando inicio a la tumorigenesis de las células de NSCLC 8.
Las tres principales vías de señalización posteriores activadas por EGFR son las proteínas quinasas activadas por mitógenos (MAPK)/ quinasas reguladas por señales extracelulares (ERK), la fosfatidilinositol 3-quinasa (PI3K)/Akt/ mTOR y la interleucina 6 (IL-6)/Janus quinasa (JAK)/transductor de señal y activador de las vías de señalización de la transcripción 3 (STAT3) 8.
Es de suma importancia identificar un nuevo blanco molecular en el cáncer de pulmón de células no pequeñas debido a que diversas alteraciones genéticas como EGFR, ALK, ROS1, RET, BRAF V600E, MET Exon 14 y NTRK requieren de una nueva posible diana molecular para el posible tratamiento, dado que la mayoría de las modificaciones genéticas representa entre un 10% y 30% albergan mutaciones activadoras en el dominio tirosina quinasa del gen EGFR en las neoplasias de NSCLC, por lo que los nuevos casos pueden elevarse hasta el 60% en personas provenientes de países asiáticos 9.
La búsqueda de una diana molecular específica se justifica ante la creciente evidencia de la resistencia adquirida a las terapias estándar y la necesidad de optimizar la precisión terapéutica. Dicha identificación no solo podría ofrecer alternativas viables para aquellos pacientes que no responden a las terapias convencionales, sino que también podría facilitar una mayor personalización del tratamiento, aprovechando las características moleculares únicas de cada tumor 4.
En el tratamiento contemporáneo del cáncer de pulmón de células no pequeñas (CPCNP), se emplean diversas moléculas diseñadas para inhibir selectivamente vías de señalización cruciales implicadas en la patogénesis de la enfermedad. Destacan entre ellas los inhibidores de tirosina cinasas, tales como Erlotinib, Gefitinib y Osimertinib, que se dirigen específicamente a mutaciones presentes en el receptor del factor de crecimiento epidérmico (EGFR). Además, se han introducido inhibidores de ALK, como Crizotinib y Ceritinib, dirigidos a tratar alteraciones en la quinasa del linfoma anaplásico (ALK). Otros fármacos como los inhibidores de MEK, entre los que se incluye el Trametinib, y los inhibidores de m-TOR, han sido incorporados al tratamiento terapéutico, ampliando así las opciones de tratamiento y proporcionando alternativas para los pacientes 10-12.
Los blancos que actúan dichos inhibidores en las tirosinas-quinasas son EGFR, ALK y ROS1, estos se encuentran alterados en el cáncer. En los inhibidores de MEK, su blanco principal es el gen BRAF, especialmente cuando está mutado. Mientras que los inhibidores m-TOR actúan principalmente en la vía de señalización PI3K/ AKT/mTOR. Los inhibidores de ciclina se dirigen a las quinasas dependientes de ciclinas (CDK) y los inhibidores de PARP cuyo objetivo es la enzima PARP, son esenciales para la reparación de daños en el ADN de una sola hebra 13.
Investigaciones recientes indican que la mediana de supervivencia global en pacientes tratados con Erlotinib fue de 30,8 meses, y la mediana de la supervivencia libre de progresión fue de 11,4 meses. Estos resultados sugieren que Erlotinib puede proporcionar un beneficio significativo en términos de tiempo antes de que el cáncer progrese 13.
Las dianas moleculares podrían mejorar la situación mostrando una mayor especificidad y eficacia del tratamiento. Este enfoque terapéutico podría minimizar los efectos secundarios y podría mejorar la calidad de vida del paciente. Por lo tanto, la identificación de biomarcadores predictivos podrían mejorar las terapias dirigidas elevando una respuesta y supervivencia favorable 14,15.
En la identificación de una diana molecular, el pipeline bioinformático es de gran utilidad en la búsqueda de posibles dianas terapéuticas en el cáncer de cáncer de pulmón de células no pequeñas. Las tecnologías en las que se basa el pipeline bioinformático son las ómicas, que incluyen la genómica, transcriptómica y proteómica. Estas técnicas permiten el análisis de los marcadores de ADN, las transcripciones de ARN y las proteínas, mediante este estudio se puede descubrir nuevos biomarcadores para aplicaciones en el pronóstico, diagnóstico y tratamiento del NSCLC16.
El objetivo del presente estudio es analizar una posible diana molecular terapéutica en la neoplasia de pulmón de células no pequeñas a través de un estudio in silico.
II. MATERIALES Y MÉTODOS
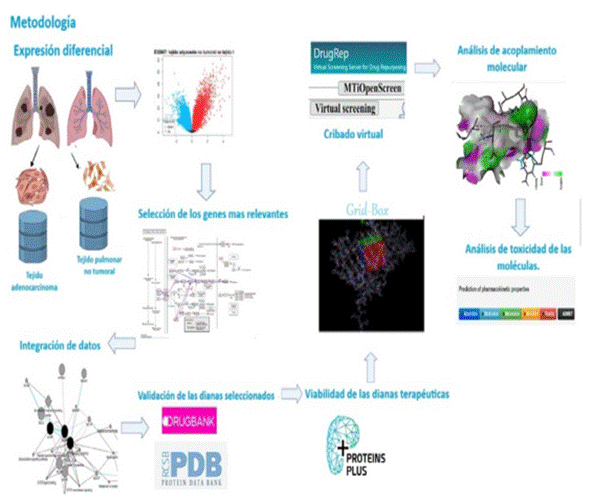
Figura 1 Metodología empleada en el estudio de una nueva diana molecular en el cáncer de pulmón de células no pequeñas.
La Figura 1 representa el procedimiento respectivo que se llevó a cabo en nuestra investigación con la finalidad de identificar el propósito de este estudio.
Expresión diferencial de los genes que sobreexpresan en el cáncer de pulmón de células no pequeñas.
Se utilizaron los criterios establecidos por Dong et al. 16 para la búsqueda de conjuntos de datos. Estos criterios incluyen:
Seleccionar conjuntos que estén enfocados en el cáncer de pulmón de células no pequeñas.
Los conjuntos de datos deben estar respaldados por publicaciones en bases de datos confiables.
Aplicando estos criterios, se accedió a la base de datos GEO (Gene Expression Omnibus) y se indagó el término "non small cell lung cancer". Después, se seleccionó: el uso de GEO DataSets, la especie Homo sapiens, experimentos que implican la generación de perfiles de expresión mediante arrays, y se examinó el diseño experimental que comparó muestras de pacientes normales con pacientes que padecen adenocarcinoma de pulmón.
Análisis de expresión diferencial
Se utilizó GEO2R en la base de datos GEO en el análisis de expresión diferencial. Se realizó diagramas de tipo volcán (Volcano plots) a través de la comparación de muestras de personas con adenocarcinoma y personas sanas. En este mecanismo se identificó los genes que están sobreexpresados en la neoplasia maligna de pulmón.
Identificación de dianas terapéuticas
Base de datos
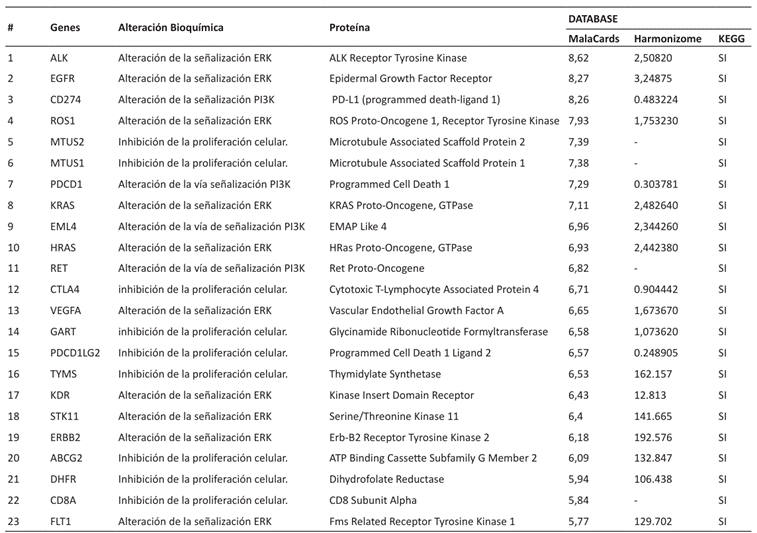
En bases de datos como Malacards, se realizó una búsqueda enfocada en genes asociados con el cáncer de pulmón de células no pequeñas. La entrada más relevante identificada para su inclusión en la investigación fue "Lung Non-Squamous Non-Small Cell Carcinoma". Esta entrada específica destaca por incluir un conjunto de 23 genes de élite relacionados con la enfermedad y un MIFTS de 55.
En Harmizone se escogió el apartado “Gene Sets" usando el término "Lung Non-Squamous Non-Small Cell Carcinoma". El objetivo de realizar este proceso fue diferenciar y analizar similitudes y congruencias genéticas con la base de datos Malacards. Se eligió los genes que tienen interacciones y genes que obtuvieron las puntuaciones más altas para el respectivo análisis.
Finalmente, se extrajo de la base de datos KEGG información sobre las vías de señalización asociadas con el cáncer de pulmón de células no pequeñas, así como las rutas metabólicas implicadas en esta enfermedad. Se recopiló información sobre los genes involucrados en las cascadas que inducen alteraciones bioquímicas y desregulaciones durante el desarrollo de la carcinogénesis del cáncer de pulmón de células no pequeñas.
Selección de los genes más significativos.
Se centró en obtener las puntuaciones estándar de cada uno de los genes disponibles en bases de datos como Malacards y Harmonizome, considerando la relación de cada gen con la patología. De la misma manera, se extrajo información sobre las alteraciones bioquímicas.
Después de la revisión, se escogió los genes más relevantes para el cáncer de pulmón de células no pequeñas. Se clasificó las alteraciones bioquímicas y sus puntuaciones. Aunque se identificaron diversas alteraciones bioquímicas en los genes, se utilizó el score obtenido para priorizar aquellos genes con puntuaciones más altas, centrando el análisis en aquellos con mayor score. Además, se enfocó en el análisis de la vía de señalización ERK (quinasa regulada por señales extracelulares).
Integración de redes moleculares
El programa Cytoscape facilitó la creación de redes biológicas, empleando el plugin GeneMania para establecer una red de interacciones entre los genes más representativos relacionados con la enfermedad. Luego, se escogieron los genes que presentaron el mayor número de interacciones en los nodos, así como las puntuaciones más altas. Además, se revisaron los bordes para identificar los genes con mayor grado de interacción.
Verificación de las dianas terapéuticas
La base de datos de DrugBank ofrece información sobre dianas terapéuticas y medicamentos relacionados con el cáncer de pulmón de células no pequeñas. Se llevó a cabo una búsqueda de los genes seleccionados anteriormente para identificar posibles opciones de ligandos. Para esta base de datos, el control de calidad, se tomó en cuenta si ya está realizada en forma target y en qué fase de tratamiento está ligada. Como resultado, se encontraron varios fármacos en desarrollo y en fase experimental.
A continuación, se encontraron en Protein Data Bank los cristales de las proteínas de los genes y se consideraron los parámetros del control de calidad para validar las dianas, los cuales son R-value Work y del R-Free, los cuales no deben exceder 0.3 17. Sin embargo, es importante señalar que en algunos casos no se localizaron todos los cristales, por lo que se recurrió al modelado in silico utilizando AlphaFold, respaldado por alineamientos estructurales y de secuencia con proteínas homólogas 18.
Evaluación de viabilidad de los objetivos terapéuticos
En el sitio web de Protein Plus de la Universidad de Hamburgo y ChEMBL del European Bioinformatics Institute (EBI), se determinó la capacidad de los fármacos para interactuar con las proteínas obtenidas. El código PDB de la proteína fue mapeado a través de la web. No obstante, si la proteína se obtuvo de AlphaFold, solo se debe cargar el archivo en formato PDB en DoGSiteScorer. Los posibles sitios y sub-sitios de unión proteica fueron identificados utilizando DoGSiteScorer.
Exploración de moléculas que podrían interactuar con los objetivos terapéuticos.
Los cristales validados en formato PDB se obtuvieron de la página web Protein Data Bank. Se eliminaron compuestos no proteicos, como el agua y los complejos, utilizando el software Chimera 1.16. Los posibles ligandos que se utilizarán fueron descargados de la plataforma PubChem. Con la herramienta Avogadro, se logró reducir la energía para obtener un acoplamiento óptimo. Las dimensiones de la Grid-Box y la ubicación del sitio de unión con los fragmentos de aminoácidos se calcularon mediante Autodocktools. La Grid-Box se colocó dentro del área de dominio más probable para facilitar la interacción entre la proteína y el ligando.
Cribado Virtual
La web DrugRep proporcionó el análisis de la detección virtual para la reutilización de medicamentos. Por lo cual se escogió los medicamentos aprobados por la FDA. Se subió la proteína en formato pdb, luego se agregó en más parámetros el número de cavidades de diez para la detección de los pockets. Se contrastó los resultados obtenidos anteriormente con la web MTiopenscreen, donde hace énfasis en las coordenadas de la GRIDBOX para obtener los ligandos de mayor energía de unión, es por ello que se basó en la elección de la librería de Druglib.
Interacción Molecular
Se empleó el software AutoDockTools para desarrollar el acoplamiento molecular rígido. Primero, se añadieron hidrógenos polares a la proteína, luego se eliminaron los hidrógenos no polares y se agregó la carga de Kollman, tras lo cual se guardó en formato pdbqt. Después, se cargó el ligando en el software, se añadieron hidrógenos y se eliminaron los no polares. Se incorporaron todos los hidrógenos y se eliminaron nuevamente los no polares. Posteriormente, se aplicó la carga de Gasteiger y se guardó en formato pdbqt.
Se optó por la modalidad de acoplamiento rígido para incorporar la proteína y se seleccionó el ligando, lo que permitió guardarlo en formato dpf. Se evaluaron las conformaciones de unión y se eligió la que presentaba la mejor afinidad para ser almacenada en formato pdb. El software Discovery Studio facilitó una comprensión más profunda del acoplamiento molecular.
Evaluación de toxicidad de las moléculas.
El análisis de las características de las moléculas fue con la web pkCSM permitiendo así conocer el análisis de toxicidad. Se realizó pruebas ADMET (Absorción, Distribución, Metabolismo, Excreción y Toxicidad) utilizando los códigos SMILES de las moléculas identificadas.
III. RESULTADOS Y DISCUSIÓN
Expresión diferencial de genes.
El análisis de expresión diferencial de genes (DEG) se aplicó con el propósito de investigar los patrones de expresión genética y desentrañar los mecanismos biológicos vinculados a enfermedades complejas. Los perfiles de expresión genética se caracterizan por la presencia de un extenso conjunto de genes (decenas de miles) y conexiones sólidas entre ellos 19.
En este estudio particular, se analizó el DEG de perfil de expresión de metilación del ADN que integra 59 muestras de tejidos de adenocarcinomas pulmonares vs tejido no tumorales adyacentes de 30 muestras con complementación de 29 muestras que eran de fumadores corriente, todo ello basado en el perfil de expresión génica de GEO: GSE32867 20.
En la Figura 2, se logró determinar el cambio de pliegue de metilación del ADN, tanto en tejido no tumoral como en tejido canceroso. En este cambio, se obtuvo 709 genes metilados diferencialmente, lo cual al ser ajustado con un valor-p de 0.05 se logró expresar 349 genes, en lo cual 164 genes están sobreexpresados de forma de color rojo lo cual son mayores a la condición de tejido no tumoral del pulmón. Mientras que solo 57 genes son de forma hipometilados que puede estar asociado con una disminución en la actividad génica, por lo cual puede considerarse de forma inhibida como están de forma de color azul 20.
El DEG que se analizó se basa en la investigación de Remmelink et al. 12 lo que sugiere que 164 genes participan en varias rutas de señalización, lo cual se logró distinguir la vía de señalización del ciclo de células de la vía de señalización de MAPK, esto debido a que se ha presentado una sobreexpresión en los genes de las familias MAPK y de la familia RAS.
Los genes KRAS, NRAS y HRAS conforman la familia RAS en los individuos, codifican cuatro proteínas similares: KRAS4A/B, NRAS y HRAS. La tumorigenesis está regulada por las proteínas RAS 21. El gen RAS codifica una familia de proteínas GTPasas que contiene a KRAS, HRAS y NRAs. El gen KRAS se encarga en codificar la proteína K-RAS, esta proteína participa como un interruptor que regula la división celular, controla la formación, maduración y la destrucción celular 22,23.
El gen HRAS codifica la proteína H-Ras, que es una diminuta GTPasa que pertenece a la familia de las proteínas RAS. Estas proteínas cumplen un rol importante en la señalización celular y regulación de mecanismos como la diferenciación, supervivencia y proliferación celular 24.
Como resultado de los genes más específicos para analizar en este estudio del DEG se obtuvo que el gen KRAS sufrió un cambio de pliegue alto en tejido canceroso en comparación del tejido normal. Otro gen específico que se encontró para el análisis es el gen EGFR, ya que se encontró de manera inhibida en su funcionalidad en tejidos no tumorales de fumadores corriente 20.
Finalmente, como base de este estudio se va tomar como eje principal el gen KRAS y este tiene correlación con el gen HRAS por pertenecer a la familia de genes RAS y a la vez el gen HRAS presentó una sobreexpresión de cambio de pliegue. La mutación de este gen conduce a proliferación celular y resistencia a la apoptosis, por lo que es importante analizar la vía MAPK/ ERK sobre los procesos de carcinogénicos y el desarrollo de novedosas dianas terapéuticas.
Nota:
Análisis de las bases de datos
La base de datos KEGG permitió identificar diferentes vías moleculares que participan los genes en el cáncer de pulmón de células no pequeñas, por lo cual, al correlacionar con el análisis de expresión diferencial, se observó que la vía de señalización ERK cerca del 20 al 30% de adenocarcinomas relacionado al cáncer de pulmón de células no pequeñas es el resultado de una alteración en el gen KRAS 15,25.
La Figura 3 se utilizó como referencia para identificar las primeras manifestaciones anormales del tejido pulmonar, como la hiperplasia y displasia. Por lo tanto, la vía de señalización MAPK/ERK se presenta en las etapas iniciales de la enfermedad, analizando los segmentos enmarcados en color morado. En la Figura 3 se observa los principales genes responsables de provocar la enfermedad, es decir el cáncer de pulmón de células no pequeñas; se representa en tonalidad roja. Las alteraciones biológicas en la vía MAPK/ERK promueven la proliferación celular descontrolada, evaden la apoptosis, permiten la angiogénesis y facilitan el desarrollo de la metástasis. Asimismo, desarrolla la resistencia a tratamientos dirigidos y modula el microambiente tumoral para promover el crecimiento tumoral. Es por ello que las funciones de esta vía MAPK/ERK tiene un objetivo clave para desarrollar nuevas terapias dirigidas al cáncer de pulmón de células no pequeñas 26. Luego que se obtuvo la ruta, se contrastó con la ayuda de la base de datos HARMONIZOME y MALACARDS para la obtención de genes de élite que participan en la carcinogénesis de pulmón.
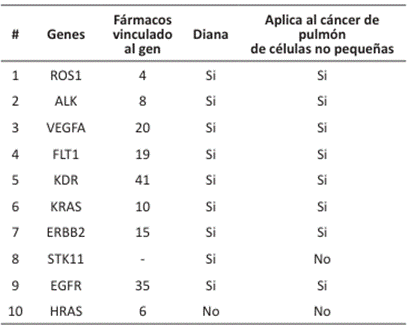
Se eligieron 23 genes de élite identificados en la base de datos MALACARDS como punto de partida para el estudio de la patología. La selección de estos genes se basó en las interacciones analizadas que relacionan los genes con la enfermedad, y cada uno de ellos cuenta con un score que indica su incidencia asociada a la enfermedad. Para este análisis, se utilizó la base de datos HARMONIZONE, que complementó la información de MALACARDS. Según los datos de la Tabla 1, los genes se agruparon en diferentes alteraciones bioquímicas (disfunciones), y se estableció un rango para seleccionar los genes implicados en la enfermedad con un valor superior a 0.5 en HARMONIZONE, debido a la falta de evidencia experimental que conecte estos genes con la enfermedad. Esta información fue verificada en la base de datos GENECARDS.
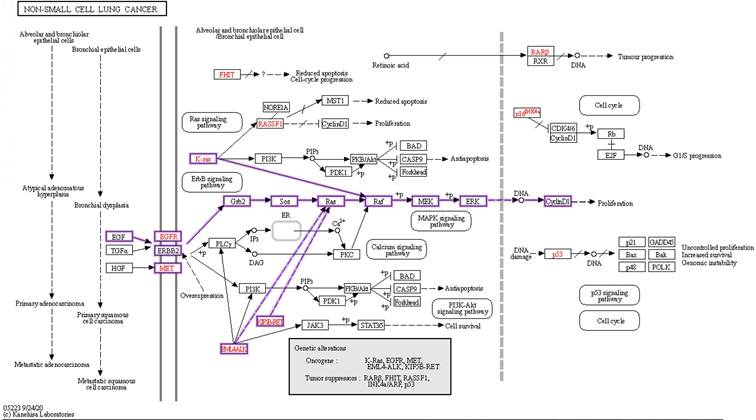
Figura 3 Análisis de la ruta de señalización del cáncer de pulmón de células no pequeñas realizado con KEGG (Kyoto Encyclopedia of Genes and Genomes).
Nota: Conjunto de genes corriente arriba en la vía MAP/ERK:
EFG: Factor de crecimiento epidérmico, EGFR: Receptor del factor de crecimiento epidérmico, ERBB2: Receptor Erb-B2 tirosina quinasa 2, MET: Receptor tirosina quinasa, GRB2: Proteína 2 unida al receptor del factor de crecimiento, SOS: Factor de intercambio de nucleótidos de guanina Ras/Rac, RAS: Rat Sarcoma, RAF: Protooncogén, serina/treonina quinasa, MEK: Proteína quinasa activada por mitógenos, ERK: Quinasa regulada por señales extracelulares, CyclinD1: Gen CCND1 - Cyclin D1.
Abreviatura del conjunto de genes corriente abajo que están presentes en la vía MAP/ERK:
EML4-ALK es una combinación génica que involucra la adhesión de fragmentos procedente de dos genes distintos: EML4 (Echinoderm Microtubule-associated protein-Like 4) y ALK (Anaplastic Lymphoma Kinase). Esta fusión da lugar a la creación de un gen de fusión que codifica una proteína quimérica, que es constitutivamente activa y promueve el crecimiento celular anormal.
IF5B-RET se refieren a una fusión génica entre los genes KIF5B (Kinesin Family Member 5B) y RET (Rearranged during Transfection).
Evaluación de las redes moleculares
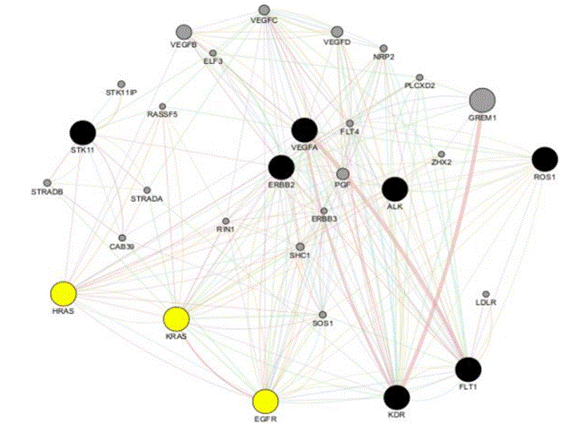
Según los resultados obtenidos, se identificó la vía de señalización ERK como la principal alteración bioquímica. Este análisis de integración de datos, se llevó a cabo utilizando el software CYTOSCAPE, que facilitó la representación de la interacción genética en cuatro niveles. Los genes ingresados en el software están destacados en color negro, mientras que los genes relacionados, añadidos a través de GeneMania, se muestran en gris. Las conexiones de diferentes colores representan las interacciones que enlazan los nodos (genes).
Vía de señalización MAPK/ERK.
En los tumores malignos de pulmón de células no pequeñas (NSCLC), la vía de señalización MAPK/ERK (Quinasa Activada por Mitógenos/ Quinasa Regulada por Señal Extracelular) se encuentra desregulado producto de alteraciones o sobreexpresiones de componentes de la familia de las RAS. La Figura 4, se destaca la interacción entre los genes que participan en la vía de señalización MAPK/ERK. Esta vía, involucra diferentes genes, por lo cual en este estudio se seleccionaron a los genes que mayor interacción presenta, es por ello que, que están representados en tono amarillo. A continuación, se describe cómo actúan los genes que tienen mayor incidencia en la vía.
Como uno de los principales genes que participa en la vía de MAPK/ERK, es el gen EGFR el cual asume la función de iniciador en la vía de señalización de MAPK/ERK. Este gen codifica el receptor del Factor de Crecimiento Epidérmico (EGFR), clasificado como un receptor tirosina quinasa (RTK). La activación de EGFR se produce al unirse a ligandos como el Factor de Crecimiento Epidérmico (EGF) o el Factor de Crecimiento Transformante alfa (TGF-α). Además, el gen ROS1 se asocia a la vía de señalización por la activación del gen, esto ocurre cuando se une a su ligando. Luego esta unión induce a la autofosforilación en residuos de tirosina en su dominio intracelular. Estos sitios fosforilados de ROS1 se unen las proteínas adaptadoras como son GRB2 y SOS, estas, proteínas son cruciales para la transducción de señales hacia abajo en la vía de señalización intracelular 27,28.
La interacción del gen VEGFA se genera a partir de la unión a su receptor especifico VEGFR-2, ubicado en las células endoteliales. La unión desencadena su activación a través de la fosforilación de restos de tirosina intracelular. Las proteínas adaptadoras Shc y GRB2 se asocian a sitios fosforilados de VEGFR-2. La proteína adaptadora GRB2 tiene un rol esencial en la activación de la cascada MAPK/ ERK y se asocia con la proteína SOS conduciendo a activar RAS, proteína importante en la vía MARPK/ ERK 29.
La activación de la serina/treonina quinasa RAF se da mediante la proteína RAS, encargada en fosforilar y activar la quinasa de doble especificidad MEK. MEK fosforila y activa la quinasa ERK. ERK se transloca al núcleo de la célula, lugar donde se realiza la fosforilación de distintos factores de transcripción 30.
Finalmente, se escogió el gen HRAS para posterior análisis molecular, ya que este presente un score de interacción apto para el análisis de la vía de señalización. KRAS es un miembro de la familia RAS y actúa como un interruptor molecular en la vía de señalización MAPK/ERK. En la cascada de activación MAPK/ERK, se activa mediante la unión a GTP a través de complejas interacciones con proteínas reguladoras lo que estimula la activación 31.
Verificación de los genes seleccionados
Se identificó varios genes asociados con el cáncer de pulmón de células no pequeñas, los cuales también están vinculados a medicamentos empleados en el tratamiento de diversas enfermedades. Para confirmar la existencia de dianas farmacológicas para los genes seleccionados, se realizó una búsqueda en la base de datos Drugbank. La Tabla 3 presenta los genes de élite seleccionados junto con los medicamentos asociados a estos genes y su posible uso en el tratamiento del cáncer de células no pequeñas. Es importante señalar que algunos de estos medicamentos también se utilizan en tratamientos para condiciones distintas al cáncer de pulmón de células no pequeñas. Se debe tener en cuenta la falta de investigación extensa en la comunidad científica sobre estos fármacos, ya que muchos aún se encuentran en fases experimentales sin una validación amplia. Además, se ha identificado que algunos de estos medicamentos podrían actuar como ligandos para el acoplamiento molecular, lo que permitirá comparar los resultados con el cribado virtual descrito más adelante 32.
Basado en la Tabla 3, los genes clave como VEGFA, FLT1, KDR, KRAS y EGFR tienen dianas terapéuticas específicas para el tratamiento del cáncer de pulmón de células no pequeñas. Estos genes son importantes porque su alteración puede agravar el desarrollo del cáncer. Por otro lado, para el gen ROS1 se ha establecido el fármaco Entrectinib, que está en fase 3 de tratamiento y también se emplea en otros tipos de cáncer, como el de colon, páncreas y ovario. En cuanto al gen ALK, cuenta con dianas específicas, siendo un ejemplo Crizotinib, un medicamento destinado al tratamiento de las alteraciones genéticas que contribuyen al desarrollo del cáncer de pulmón de células no pequeñas (drugbank) 33,34.
En la base de datos de Drugbank no se encontró información para el gen STK11, lo que sugiere que aún se encuentra en fase experimental. De manera similar, el gen HRAS tiene medicamentos en fase experimental, y estos aún no se utilizan en tratamientos. En este estudio, se analizó las dianas de genes que no estén directamente relacionadas con la enfermedad. Por ello, se seleccionó el gen HRAS debido a la información disponible sobre un posible fármaco, que se utilizará como referencia para comparar con los resultados obtenidos en el cribado virtual, como se mostrará más adelante.
Análisis de factibilidad de las dianas terapéuticas
Se identificó el sitio objetivo más importante de las proteínas mediante un fármaco que tiene una similitud a la estructura tridimensional de un ligando. El análisis del gen HRAS se realizó a través de la herramienta DoGSiteScorer proveniente del servicio web Protein Plus. Los sitios de unión se detectaron mediante esta herramienta computacional, donde se caracterizó la estructura, tamaño y propiedades químicas de las cavidades previstas de la proteína. Se priorizó el análisis del gen HRAS debido a la falta de información sobre fármacos en fases de tratamiento del cáncer en la base de datos DrugBank.
La herramienta DoGSiteScorer facilitó la identificación de áreas en la superficie de la proteína donde es favorable la colocación de un objeto esférico, como se describe en el estudio de Volkamer et al. (35). DoGSiteScorer clasifica estas ubicaciones en subbolsillos basándose en un umbral de densidad, detallando los subbolsillos según la cantidad de átomos o grupos funcionales y la composición de aminoácidos, proporcionando así información sobre las propiedades fisicoquímicas de la cavidad. Además, se examinó la lipofilia de estas cavidades mediante el análisis de la superficie lipófila y la evaluación de la hidrofobicidad global. Si se dispone de un ligando, también se calcula la superposición entre el ligando y el volumen del bolsillo, según lo mencionado por Fährrolfes et al. 36.
El código de la proteína obtenida en la base de datos PDB es 1GNR, lo que permitió obtener la estructura tridimensional de la proteína HRAS. La capacidad de la proteína para que pueda ser "drogable" se evaluó mediante DoGSiteScorer. La escala que va desde 0 se considera baja drogabilidad, mientras que 1 se considera como máxima drogabilidad. Una puntuación que comprende entre 0.5 y 1 significa que una proteína es potencialmente un objetivo terapéutico. En nuestro caso, la proteína seleccionada obtuvo una puntuación de 0.77, lo que se considera un objetivo terapéutico de alto valor debido a su gran drogabilidad 37.
Para el análisis de la drogabilidad, se seleccionó el "pocket_0" por su mayor potencial para la interacción con fármacos. En la Figura 5 se muestra la estructura tridimensional de la proteína HRAS, con un volumen de cavidad de 535,4 ų y una profundidad de 586,46 Ų Estos bolsillos incluyen parámetros como profundidad, volumen, rango de drogabilidad y composición de aminoácidos. Según Fitri et al. 38, un bolsillo con gran volumen, amplia profundidad y alta proporción de aminoácidos apolares es efectivo para evaluar la capacidad de interacción con fármacos.
Cribado virtual
Para evaluar el cribado virtual, se utilizó la web Drugrep, la cual se basa en descubrir que fármaco es óptimo en el acoplamiento de la proteína que se subió a la web, es por ello
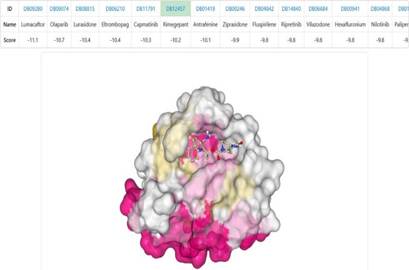
que en los resultados obtenidos se clasificó según las puntuaciones de acoplamiento o las puntuaciones de similitud, lo que permite exhibir información sobre los fármacos, junto con las interacciones de los aminoácidos que existen entre el ligando y la proteína en 3D (Figura 6).
Para contrastar los resultados de Drugrep, se utilizó MTiOpenScreen, que predice la interacción entre fármacos y ligandos ofreciendo datos sobre la afinidad de unión de las proteínas con los ligandos. En este análisis, se cargó la estructura de la proteína HRAS y se empleó la biblioteca de fármacos comerciales Drugs-lib. Las coordenadas de la Grid-Box, obtenidas previamente en DogSiteScorer y que identificaron el pocket_0 con los aminoácidos residuales, fueron transferidas a AutodockTools.
En MTiOpenScreen, se obtuvieron 7173 ensayos de interacción entre proteínas y ligandos provenientes de Drugs-lib, específicos para cada proteína seleccionada (figura 6). Se clasificaron los 100 mejores resultados basados en la afinidad de interacción y se seleccionaron los que presentaron las mejores energías de interacción. De estos, se eligió el fármaco Rimegepant, ya que apareció tanto en Drugrep como en MTiOpenScreen. Cabe destacar que no se escogieron los primeros fármacos listados en Drugrep debido a que, en las pruebas ADMET, presentaron resultados positivos en hepatotoxicidad.
Análisis de docking molecular rígido
Se llevó a cabo una investigación documental acerca de las dianas terapéuticas que están predispuestas para el gen HRAS, esto con el objetivo de tomar como control y luego comparar su afinidad de unión con el obtenido que es el medicamento Rimegepant obtenido de drugrep y MTiopenscreen. Es por ello que en el gen de este estudio se tomó como control el ligando del estudio de Salem et al. 39, donde menciona el uso de ligandos sintetizados derivados que contiene pirimidine-2-thione con fracciones incorporadas con diferentes heterociclos como la pitroazolina, la pirazolina y la pirimina. De estas tres fracciones, la afinidad de unión es de -7.9 kcal/mol, -8.8 kcal/mol y de -11.16 kcal/mol.
En nuestro estudio se seleccionó el fármaco Rimegepant debido al contraste de utilizar las herramientas de cribado virtual, las cuales fueron MTopenscreen y Drugrep; estas permitieron obtener el medicamento mencionado. Este medicamento actúa como un antagonista, ya que podría inhibir la vía MAPK/ERK. En el análisis de acoplamiento molecular para la proteína HRAS se obtuvo una afinidad de 13.02 kcal/mol para su posterior análisis de toxicidad de la molécula. En la Figura 7 se pueden ver los aminoácidos residuales en las interacciones entre el ligando y la proteína, los cuales fueron analizados utilizando el software Discovery Studio, que también facilitó la obtención de visualizaciones en 2D y 3D.
Este medicamento no es específico para el NSCLC, ya que el Rimegepant participa como receptor del péptido asociado con el gen de la calcitonina (CGRP), por lo que actualmente no forma parte de una terapia específica contra el NSCLC, pero a través del análisis del acoplamiento molecular rígido proporciona información valiosa para reutilizar el medicamento para el tratamiento del cáncer.
La interacción entre el ligando y la proteína se analizó para identificar la afinidad de unión, comparando el fármaco control con el fármaco seleccionado durante el cribado virtual. El fármaco presentó una mayor afinidad de unión con la proteína en relación al medicamento control. Las interacciones hidrofóbicas, las fuerzas de Van der Waals, enlaces de hidrógeno y las interacciones electrostáticas son fuerzas interactivas que ocasionan una mayor afinidad 40.
Los principales factores que benefician para que tengan mayor afinidad contiene las interacciones moleculares débiles como son las interacciones hidrofóbicas y los enlaces de hidrógeno. Dichas interacciones son favorables para que los ligandos obtengan mayor energía favorable, especialmente dentro de un marco estructural en el que las proteínas adoptan una conformación abierta. Según Varma et. al 41, menciona que la afinidad de unión del ligando se aumenta mediante los enlaces de hidrógeno, ya que estos, poseen la característica de desplazar las moléculas de agua unidas a la proteína. Además, cuando las interacciones hidrofóbicas son más pronunciadas en el núcleo activo de la interfaz entre el fármaco y su objetivo, se observa un incremento adicional en la actividad biológica del fármaco 42.
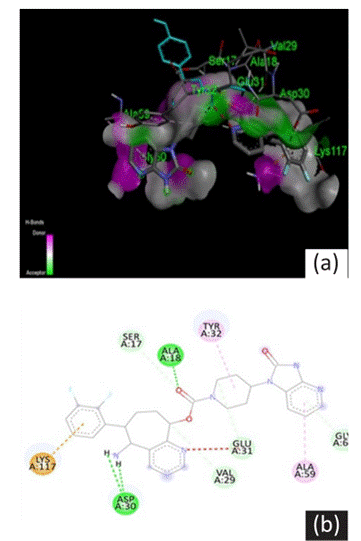
La Figura 7, muestra una conexión carbono- hidrógeno convencional en el aminoácido AlaA:18, AspA:30. Esta interacción es simple en su naturaleza, aunque su escasa reactividad en comparación con otros compuestos la convierte en un enlace robusto y resistente a la ruptura. Su formación se debe al hecho de que está compuesto por 8 electrones el átomo de carbono, lo cual es necesario para la estabilidad al adherirse con los hidrógenos cercanos, según lo señalado por Dunga et al. 43.
En el anillo de benceno forma un enlace de Pi- catión en conjunto con el aminoácido LysA:117, en este anillo aromático hay la presencia de dos átomos de fósforo incorporados, estos pueden introducir una carga positiva en la estructura (catión).
Esta interacción no covalente (Pi-catión) se forma cuando la nube electrónica Pi- del benceno interactúa con el catión formado por los átomos de fosforo 44. Otra característica que es importante mencionar es que la interacción es un resultado de desplazamiento de los electrones pi en los enlaces dobles 44. Por otro lado, la interacción no covalente Pi- Alkyl con AlaA:59 consiste en la relación de la zona hidrofóbica del ligando con el grupo alquilo presente situada en la posición 59. Esta interacción favorece la estabilidad del complejo proteína-ligando al facilitar un contacto entre la nube electrónica del Pi anillo aromático del ligando y el grupo de la Alanina.
La fuerza Pi Alkyl contribuye a la formación de una unión más robusta y puede desempeñar un papel esencial en la estructura tridimensional del sitio activo. La interacción Pi Donor Hydrogen Bond con GlyA:60 implica que el grupo amino de la glicina en la posición 60 actúa como donador de enlace de hidrógeno pi. Este comportamiento sugiere una donación de electrones a través del sistema pi, contribuyendo a la estabilización del complejo. La interacción Pi Donor hace que refuerce la especificidad de la unión al crear una conexón entre el ligando y la proteína 45,46. La interacción del GluA:31 con el nitrógeno sucede en la esquina del anillo aromático del benceno puede considerarse como una interacción desfavorable Aceptador- Donador. Esto indica que tanto el aminoácido y el nitrógeno actúan como aceptadores en la formación de enlaces de hidrogeno, lo que podría provocar una repulsión o una interacción subóptima que afecta la estabilidad del complejo ligando.
Por otro lado, la formación de un enlace pi donador entre GluA:31 y otro ciclohexano- nitrógeno implica que el grupo carboxilo de glutamato puede actuar como donador de electrones a través de su sistema pi. Este tipo de interacción pi donador puede contribuir a la estabilización del complejo proteína-ligando al favorecer la formación de enlaces más fuertes y específicos 47,48.
Nota: a) Estructura 3D de la proteína con el ligando.
b) Estructura 2D de interacciones moleculares.
Se sugiere que para un posterior análisis de estudio se debe realizar un diseño racional del medicamento, esto con la finalidad de eliminar las interacciones desfavorables que presenta dicha interacción y a la vez aprovechar los fósforos que están libres en el ligando, esto permitiría obtener una mejor afinidad de unión entre ligando y proteína.
Análisis de la toxicidad de la molécula
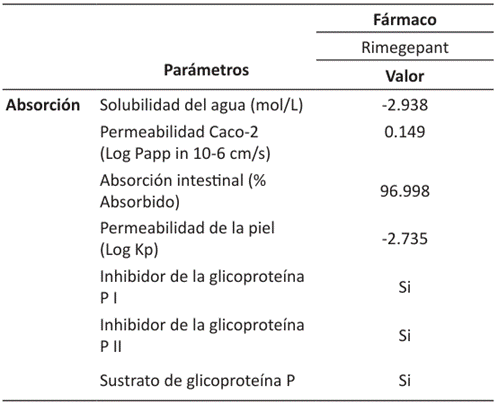
El estudio ADMET permite conocer como es la interacción del fármaco en todas las etapas que comprende la farmacocinética. Cada parámetro de ADMET permite conocer el resultado de una sustancia química al reaccionar con distintos órganos del cuerpo. La importancia de ADMET en los compuestos es primordial, en particular las sustancias químicas extrañas que se consumen a largo plazo (49). En la Tabla 5 se visualiza los parámetros que aporta la absorción del fármaco Rimegepant. La solubilidad del agua de un compuesto (logS) refleja la solubilidad de la molécula en agua a 25°C. La solubilidad en agua prevista de un compuesto se expresa como el logaritmo de la concentración molar lo que representa un valor de -2.938 log mol/L 50. La permeabilidad Caco-2 consiste en reconocer sustancias farmacológicas con una alta absorción intestinal en los seres humanos 51. Los valores predichos >0,90 representa una alta permeabilidad. Sin embargo, el valor del fármaco es de 0.149 lo que representa inferioridad a los valores predichos. La absorción intestinal está constituida por el epitelio intestinal que cuenta con varios tipos de células dispuestas en una capa columnar. Los enterocitos son las células que facilitan la absorción controlada de moléculas de la luz intestinal, incluida agua, diversos iones, nutrientes y fármacos 52. Una absorción intestinal inferior al 30% de la molécula, se considera una absorción ineficiente (50), pero como se puede observar en la Tabla 4, el fármaco es óptimo dado que posee 96.99% de absorción. Se considera que un fármaco tiene una permeabilidad cutánea muy baja, sí tiene un log Kp > −2,5. Por lo tanto, el fármaco de estudio podría ser de gran interés en el desarrollo de la administración transdermica de fármacos. La glicoproteína P cumple la función de barrera biológica al eliminar las toxinas de las células 50. En la Tabla 5 se predice si Rimegepant actúa o no como sustrato en la glicoproteína P.
El resultado es que puede actuar como sustrato y también como inhibidor de la glicoproteína P I y P II. Los sustratos de la glicoproteína P tienen la facultad de actuar como inhibidores o inductores de su función 53. Por otra parte, la glicoproteína P tiene la capacidad para expulsar sustancias provenientes del fármaco, ocasionando una disminución en la eficiencia de la terapia farmacológica 54.
En lo referente a las características que presenta en la distribución. En la Tabla 6 el
valor de la permeabilidad BBB representa si la molécula puede atravesar la barrera hematoencefálica cuando el resultado es superior a 0.3, mientras que los compuestos con un log BB inferior a -1 no tienen la capacidad de llegar al cerebro, prediciendo que Rimegepant difícilmente puede atravesar al cerebro 49. La fracción unida predice la fracción que no estará ligada en el plasma, lo que se puede observar en el resultado de la Tabla 6. La permeabilidad del SNC (sistema nervioso central) considera que los compuestos con un logPS >-2 tienen la capacidad de ingresar en el SNC, mientras que las moléculas con logPS <-3 no pueden penetrar en el SNC 55. Los compuestos activos del SNC pueden ser fatales y causar toxicidad, por lo que al tener -3.305 hace referencia que no puede atravesar hacia el SNC. Cuanto el volumen sea mayor, una mayor cantidad de fármaco se va a distribuir en los tejidos en lugar del plasma 50.
Para el tratamiento de cáncer de pulmón de células no pequeñas, la vía oral es generalmente preferida debido a su mayor biodisponibilidad sistémica, facilidad de administración, y capacidad para proporcionar un efecto terapéutico sostenido. La vía nasal puede ser limitada en términos de absorción y distribución del medicamento, y puede causar irritación local, lo que reduce su efectividad para este tipo de tratamiento.
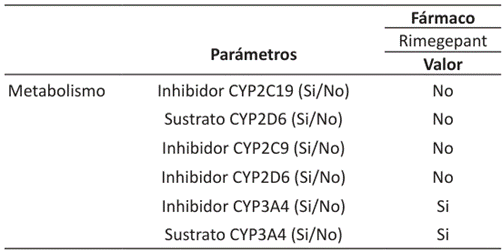
Las propiedades predictivas acerca del metabolismo de Rimegepant se presenta en la Tabla 7. Las dos enzimas que se encargan del metabolismo de fármacos P450 más prevalentes son P450 3A4 y P450 2C9, las cuales se encuentran en el hígado e intestino delgado. El citocromo P450 es una superfamilia de importantes enzimas desintoxicantes del cuerpo 56. Es posible que el fármaco no afecte la actividad de la enzima CYP450, lo que sugiere que no afectará el metabolismo ni la eliminación de los diversos sustratos farmacológicos de CYP2C19, CYP2C9 y CYP2D6 56.
El único que puede afectar es el sustrato e inhibidor de CYP3A4. Este conjunto de enzimas se encarga en degradar más del 50% de todos los fármacos administrados, y puede reducir la biodisponibilidad y eficacia terapéutica de los fármacos aumentado las concentraciones plasmáticas de los medicamentos si CYP3A4 es inhibido. Si existe inactivación de CYP3A4 ocasiona diversas interacciones farmacológicas no favorables y podría modificar la disposición, eficacia o toxicidad del medicamento 57.
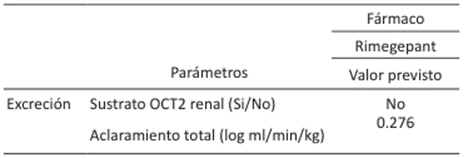
Las propiedades de excreción de Rimegepant se visualiza en la Tabla 8. OCT2 es un transportador de captación renal que desempeña un papel importante en la excreción renal de medicamentos. El fármaco no se comportará como sustrato de OCT2. El aclaramiento total es un parámetro que determina la excreción de una molécula mediante el paso hepático y renal como una combinación para determinar constante proporcionalidad del aclaramiento total 55. La eliminación total prevista de las Rimegepant se da en log(ml/min/kg), siendo el valor de 0.276.
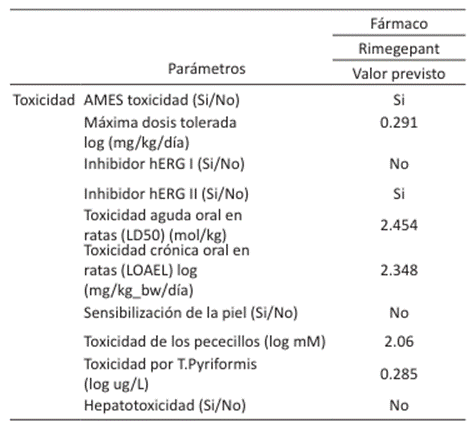
De la Tabla 9, la predicción de ADMET es positivo en este estudio. La máxima dosis tolerada (MRTD) proporciona una estimación de la dosis tóxica. Un compuesto se clasifica como de baja toxicidad si su MRTD es menor o igual a 0,477 log (mg/kg/ día), y como de alta toxicidad si supera ese valor. El valor 0.291 estima un valor bajo, lo que significa una baja toxicidad de Rimegepant. La predicción indica que no hay posibilidad que sea inhibidor de hERG I, pero para el caso de hERG II, si existe probabilidad. Los valores de dosis letales (LD50) son una medida estándar de toxicidad aguda y se define como la cantidad de un compuesto que causa la muerte de 50% de un conjunto de animales de experimentación, y se miden a través de los índices ORAT y ORCT donde los valores se reportan en mol/kg. En este caso, el valor es de 2.454 mol/kg. El valor de la toxicidad crónica oral en ratas (LOAEL) es de 2.348 log (mg/kg_bw/día) y tiene como objetivo reconocer la dosis mínima de un compuesto que causa un efecto adverso observable.
La sensibilización de la piel es negativa, lo que indica que no hay asociación de generar alergia dérmica. La toxicidad de los pececillos es un parámetro que se encarga en medir si la concentración de una molécula puede causar la muerte del 50% de estos animales. El resultado muestra un valor de 2.06 mM, lo que se considera como toxicidad aguda mínima, ya que supera los 0.5 mM. T. piriformis, un protozoo bacteriano, se utiliza frecuentemente para evaluar la toxicidad. El valor esperado para este parámetro es 0.285 log (ug/L). La hepatotoxicidad por fármacos es un problema de seguridad para el desarrollo de nuevos fármacos 50. Las lesiones hepáticas se asocian con las anomalías en el hígado, por lo que se deduce que el fármaco es negativo para causar toxicidad hepática.
Rimegepant puede ser de utilidad en el posible tratamiento de neoplasias malignas de pulmón de células no pequeñas acorde los hallazgos que muestra esta investigación. Los resultados obtenidos permitirán avanzar en investigaciones futuras donde pueda tener un rol determinante en la curación y mejoría del cáncer de pulmón de células no pequeñas en pacientes que padecen esta patología.
IV. CONCLUSIONES
En el análisis de la expresión diferencial, se encontró una sobreexpresión en el tejido canceroso, particularmente en las rutas de señalización del ciclo celular y MAP/ERK. El gen KRAS mostró un cambio significativo en el tejido tumoral, mientras que el gen EGFR fue inhibido tejidos no tumorales de fumadores. Además, se identificó una relación entre KRAS y HRAS siendo este ultimo relacionado con la proliferación celular y la resistencia de la apoptosis.
Estos hallazgos, permiten un énfasis en analizar la vía MAPK/ERK como posible diana terapéutica en el desarrollo de terapias contra el cáncer pulmonar.
La vía de señalización MAPK/ERK mediada por alteraciones en genes clave como KRAS, destaca como un elemento crucial como interruptor molecular, por lo cual al desencadenar dicha cascada destaca como función iniciadora del gen EGFR. La activación de EGFR desencadena una cascada de eventos, involucrando genes como ROS1, VEGFA, y HRAS, que se conectan en la transducción de señales hacia abajo en la vía. La relevancia entre GRB2 y SOS como proteínas adaptadoras en la activación de la cascada MAPK/ERK. Dentro de la cascada, se logró seleccionar el gen HRAS, lo cual resalta su función que desempeña como un interruptor molecular en la activación de la cascada. En el docking molecular entre el fármaco Rimegepant y la proteína HRAS se identifican interacciones hidrofóbicas, siendo una de ellas el enlace de puente de hidrógeno convencional en ALA18 y ASP30, lo que contribuye de manera significativa la estabilidad de unión del ligando-proteína. Otra interacción importante es el enlace de pi-alquilo que contribuye a la especificidad de la unión y a la vez a la potencia del fármaco, facilitando la formación del complejo ligando-diana, lo que se traduce en una mayor eficacia terapéutica.
El fármaco Rimegepant muestra resultados prometedores en el análisis ADMET debido a que no muestra hepatotoxicidad en los seres humanos y los parámetros de toxicidad son realmente favorables, lo que permitirá estudiar con más profundidad este medicamento, en estudios posteriores