1 Introducción
Según INEC y ESPAC (2016), en el Ecuador se producen 6’202.408 litros de leche diarios, en la región Sierra 4’810.551 litros, (el 75% de la producción nacional), y en la provincia de Pichincha 873.272 litros. Torres (2018) estima que el cantón Cayambe produce alrededor de 425.000 litros diarios de leche, de los cuales destinan para el consumo 51.000 litros, producción artesanal 106.250 litros y venta a granel 267.750 litros. De la misma manera, Céspedes y Pachacama (2012), afirman que en el cantón Pedro Moncayo existe una producción diaria de leche de 13 905 litros diarios.
Bonifaz y Conlago (2016) manifiestan que la mastitis causa grandes molestias a los ganaderos, provocando una disminución en la producción y características de calidad de la leche, viéndose afectada en su composición química, física y bacteriológica, presentando un menor porcentaje de sólidos totales, proteínas, grasa y calcio. Se conoce como mastitis a la inflamación e irritación de la glándula mamaria causada por diversos agentes patógenos, presentando aumento de células somáticas y provocando malestar en el animal; como efecto secundario de esta infección, la textura de la leche cambia sus características organolépticas (Bhattarai y col., 2018). Según Martínez (2012), los síntomas clínicos son aumento en el número de leucocitos, composición y apariencia alterada (grumos), fiebre, cuartos mamarios enrojecidos, hinchados y calientes.
La aparición de la mastitis depende de las condiciones higiénicas de las salas de ordeño (Zhang y col., 2018), edad, raza, partos, periodo de lactancia, producción de leche y condiciones del equipo de ordeño. En caso de que no se utilice equipo de ordeño, se deberá controlar la salud e higiene de la persona encargada del ordeño de las vacas (Ruiz Gil, Peña Rodríguez y Remón Díaz, 2016).
La enfermedad aparece también cuando los factores del medio y el manejo de los animales interactúan de tal manera que la ubre es expuesta a microorganismos patógenos. Hay tres factores esenciales que promueven la mastitis, el hospedador, el agente infeccioso y el medio ambiente (Owens y Nickerson, 2011).
Se han realizado estudios donde se identifican los agentes etiológicos causantes de mastitis (Cervantes y col., 2017); empleando medios de cultivo selectivos se han aislado Streptococcus agalactiae, Streptococcus uberis, Staphylococcus aureus, E. coli, Klebsiella, Pseudomona, Candida albicans y Proteus mirabilis.
Hernández y col. (2015) en un estudio sobre identificación de agentes etiológicos determinaron que Staphylococcus aureus es el microorganismo más importante, con un 26% de prevalencia sobre el total de microorganismos identificados en el departamento de Boyacá.
El método estándar para la identificación de los agentes patógenos consiste en aislamientos en medios de cultivos selectivos, sin embargo, usando este método solamente se hará un diagnóstico hasta el nivel de género de la bacteria (Peña y col., 2012).
La prevalencia de esta enfermedad en el cantón Cayambe y Pedro Moncayo es preocupante, pues los microorganismos patógenos responsables cambian continuamente su dinámica ecológica por las mutaciones que sufren los agentes etiológicos. Por esta causa se han tomado medidas para identificar los agentes patógenos causantes de la mastitis por medio de pruebas bioquímicas y técnicas moleculares que permiten establecer los agentes patógenos y la resistencia a múltiples antibióticos por el mal uso de estos al tratar esta enfermedad (Bonifaz y Conlago, 2016).
Con base a lo mencionado, los objetivos de esta investigación son determinar mediante técnicas moleculares los agentes etiológicos de la mastitis bovina de muestras provenientes de unidades productoras de los cantones Cayambe y Pedro Moncayo, así como también identificar las bacterias de la enfermedad mediante 3 pruebas bioquímicas, detectar los genes de resistencia blaTEM para betalactámicos y tetA para tetraciclinas, en bacterias aisladas en esta investigación mediante la técnica PCR y por último realizar la identificación molecular de los microrganismos aislados a través del secuenciamiento de la región ribosomal RNA 16S.
2 Materiales y Métodos
2. 1 Toma de muestras de leche
Las muestras de leche fueron tomadas en fincas ganaderas ubicadas en los cantones de Cayambe (coordenadas 0◦02’38”N 78◦09’22”O) y Pedro Moncayo (coordenadas 0◦02’37”N 78◦20’57”O), las cuales habían sido previamente identificadas como positivas para mastitis mediante California Mastitis Test (CMT).
Se colectaron un total de 24 muestras de leche siguiendo el protocolo LCL 001 del Laboratorio de calidad de leche de la Universidad Politécnica Salesiana, el cual indica que se deben tomar 40 mL de leche por envase, para su análisis en el equipo FOSSOMATIC células somáticas (CCS) por citometría de flujo. Se seleccionaron para este estudio aquellas muestras que superaron los 500.000 CCS/dL según lo determinado por Nieto y col. (2012).
2.2 Aislamiento microbiano
Las muestras de leche fueron sembradas por hisopado en medio Tripteína Soya Agar (TSA), recomendado para la detección y recuento de una amplia gama de bacterias, y se incubaron a 37 ◦C por 48 horas (Britania Tripteína Soya Agar, 2015). Posterior a ello, para obtener colonias puras se realizaron varios subcultivos empleando la técnica de siembra por agotamiento en medio TSA (Milián y col., 2014). Se confirmó la presencia de colonias bacterianas puras mediante tinción de Gram.
2.3 Identificación mediante pruebas bioquímicas
Se realizaron cuatro pruebas bioquímicas de identificación bacteriana: Tinción de Gram para diferenciar bacilos y cocos Gram (+) y Gram (-); Catalasa empleando peróxido de hidrógeno al 30%, esperando visualizar presencia o ausencia de burbujeo (Wanger y col., 2017); Prueba Coagulasa en plasma humano, donde se inocularon las cepas puras e incubadas a 37 ◦C por 24 horas para analizar presencia o ausencia de coagulación (American Society for Microbiology, 2016); y Agar Manitol Sal (AMS) en donde se realizaron siembras de las cepas aisladas y fueron incubadas a 37 ◦C por 48 horas con la finalidad de diferenciar bacterias del género Staphylococcus (Vila, Zhurbenko y Viera, 2004). La aplicación de estas pruebas tuvo como finalidad evitar que los análisis moleculares se realicen en especies bacterianas repetidas.
2.4 Extracción de ADN y amplificación de la región 16S rRNA
Las cepas puras fueron masificadas en medio Tryptic Soya Broth (TSB) hasta llegar a una concentración de 12×108 (UFC/mL) la cual fue determinada por comparación con patrones de turbidez de la escala McFarland. En tubos de 1,5 mL se colocó 1mL de cultivo bacteriano de cada cepa masificada y la muestra se centrifugó para formar un pellet de bacterias que fue empleado para la extracción de ADN en base a lo recomendado por Becton et al. (2005). La extracción de ADN total se realizó empleando el protocolo de extracción de ADN de Köchl, Niederstätter y Parson (2005). La presencia o ausencia de ADN fue confirmada mediante electroforesis en gel de agarosa al 1% (Buitrago, 2010). El ADN obtenido fue conservado en una solución de Tris-EDTA a -20◦ C (Tan y Yiap, 2009).
La amplificación de la región 16S rRNA se realizó usando los primers 27F(5 TCCTACGGGAGGCAGCAGT3 ) y 1492R (5 GGACTACCAGGGTATCTAATCCTGTT3 ) diseñados por Marchesi y col. (1998). Se usó la técnica PCR, utilizando el termociclador marca Labnet Multigene bajo las siguientes condiciones: desnaturalización inicial a 95 ◦C por 5 minutos, 25 ciclos de desnaturalización a 95 ◦C por 1 minuto, annealing a 60 ◦C por 2 minuto, extensión inicial por 1 minuto a 72 ◦C y extensión final de 7 minutos a 72 ◦C seguido de mantenimiento a 4 ◦C (Kang y col., 2015). Se determinó la presencia de amplicones realizando electroforesis en gel de agarosa al 1%.
2.5 Secuenciación y análisis de secuencias
Los amplicones fueron secuenciados bajo la técnica de Sanger en MACROGEN, Corea del Sur. Las secuencias obtenidas se compararon con secuencias existentes en el GeneBank del Centro Nacional para la Información Biotecnológica (NCBI).
Posteriormente, las secuencias fueron alineadas con la herramienta Muscle de software Mega 6 para obtener un árbol filogenético empleando la herramienta neighbor joining de Mega 6 para realizar una correcta clasificación taxonómica de los agentes etiológicos aislados (Fuertes y Cerna, 2018).
2.6 Amplificación de genes de resistencia a antibióticos blaTEM (β-lactámicos) y tatA (tetraciclinas)
La presencia o ausencia de genes de resistencia a antibióticos se determinó empleando los siguientes primers específicos descritos por Tao y col. (2014), para blaTEM: Forward (5 GCA CGA GTG GGTTAC ATC GA 3 ) y Reverse (5 GGT CCT CCG ATCGTT GTC AG 3 ) el tamaño aproximado del gen de resistencia es de 300 pb; para tetA: Forward (5 GCT
ACA TCC TGC TTG CCT TC 3 ) y Reverse (5 CATAGA TCG CCG TGA AGA GG 3 ). Según Belding y Boopathy (2018) el tamaño aproximado del gen de resistencia es de entre 250 a 300 pb. Los resultados obtenidos fueron verificados en electroforesis en gel de agarosa al 1%.
3 Resultados y Discusión
3.1 Identificación mediante pruebas bioquímicas
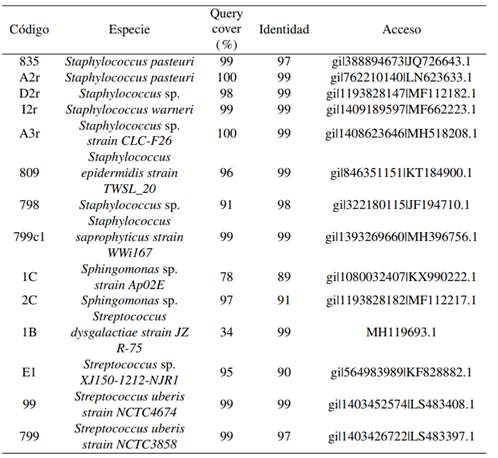
Se aislaron un total de 17 cepas bacterianas. La tinción Gram dio como resultado 15 especies Gram positivas y 2 especies Gram negativas, como se indica en la Tabla 1.
Tras la aplicación de las pruebas de catalasa se identificaron 11 especies catalasa positivo y 6 especies catalasa negativo. Las pruebas de coagulasa permitieron identificar 1 especie coagulasa positivo y 16 especies coagulasa negativo. Chaneton (2010), afirma que los Staphylococcus coagulasa negativos son los patógenos con mayor predominio, al ser identificados en función al tipo de infección 47% en infección latente y 28% en mastitis subclínica.
Las siembras en Agar Manitol Sal (AMS) permitieron catalogar a 4 especies como positivas a fermentación de manitol, 8 especies negativas a fermentación de manitol y 5 especies bacterianas que no dieron un resultado determinado.
3.2 Identificación mediante métodos moleculares
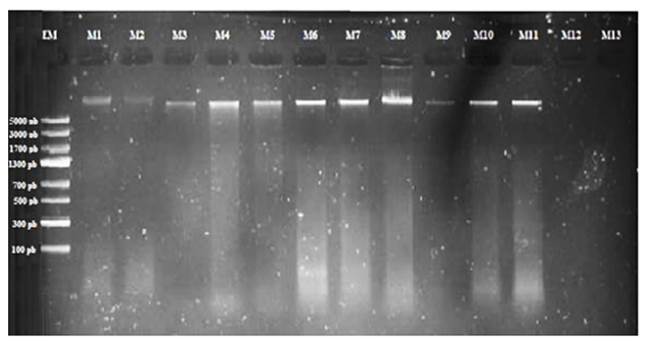
El ADN total obtenido fue de aproximadamente 7000 pb, basándose en la escalera molecular del kit Thermo Scientific 2× Phire Plant Direct (Figura 1).
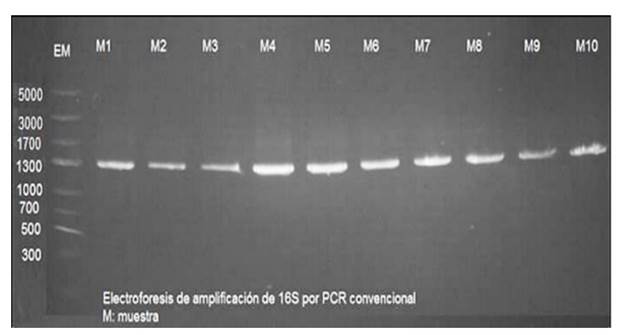
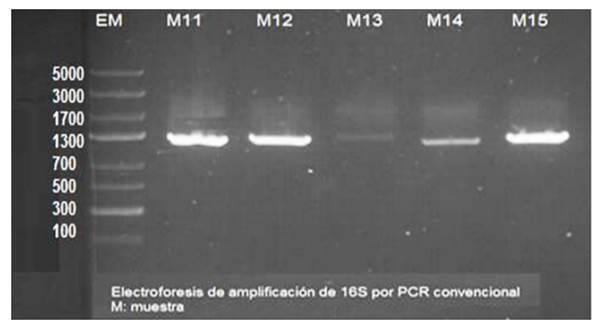
La amplificación de la región 16S rRNA dio como resultado bandas de aproximadamente 1300 pb (Figuras 2 y 3). La validez de las secuencias obtenidas fue analizada empleando el software FinchTV, con lo cual se descartaron 3 secuencias por no contar con la calidad adecuada. Con las secuencias restantes se obtuvo la identidad de 15 especies correspondientes al dominio Bacteria.
Las secuencias obtenidas en esta investigación fueron comparadas con secuencias presentes en la base de datos en el GeneBank del NCBI. Los porcentajes de identidad superaron el 97% en todos los casos, a excepción de las cepas codificadas como 1C, 2C y E1. Sol-Church y Frenck (2014), sugieren aceptar porcentajes de identidad superiores al 95%. Las secuencias de las especies catalogadas como D2r, 798 y 2C no contaron con un registro taxonómico en la base de datos del NCBI, siendo catalogadas como bacterias no cultivadas. Para realizar una correcta identificación de los agentes etiológicos aislados se elaboró un árbol filogenético empleando las 15 secuencias obtenidas y 4 secuencias externas usadas de referencia descargadas de la base de datos del NCBI (Figura 4).
Las especies aisladas fueron identificadas como agentes etiológicos causantes de mastitis en el ganado bovino lechero en la zona de estudio. Basados en la información obtenida con el árbol filogenético, se establecieron tres clados:
Clado (I): Los aislamientos identificados como Staphylococcus sp. (A3r), S. pasteuri (835, A2r), S. Warneri (I2r), S. epidermidis (809) y S. saprophyticus (799c1), se encuentran en este clado. Existieron especies que no pudieron ser identificadas a nivel taxonómico como es el caso de Bacterium strain 2BL_4 (D2r) la cual se alineó con S. warneri y una bacteria no cultivada clone ncd2254e01c2 (798), especies que se agruparon a la secuencia de referencia de Staphylococcus aureus usada como especie externa, determinando que estas pueden pertenecer al mismo género, pero de diferente cepa.
Clado (II): Incluyen a Sphingomonas sp. (C1) y Bacterium strain 4BL_5 (2C) agrupadas estrechamente con la cepa de referencia tipo de ATCC Sphingomonas paucimobilis. Esta relación la confirma Clavijo y col. (2012), ya que existen géneros que se encuentran dentro de un mismo grupo, comparten características y coinciden también con la identificación molecular.
Clado (III): Todos los aislamientos en el clado III coinciden al ser del mismo género Streptococcus al agruparse en las siguientes especies S. uberis (99, 799), S. dysgalactiae (B1) el cual se agrupó con Streptococcus sp. (E1).
En la Tabla 2 se evidencian los géneros de los agentes patógenos identificados en esta investigación, que son Staphylococcus, Sphingomonas y Streptococcus. Existen bacterias que son componentes normales de la microbiota cutánea, entre ellas bacterias del género Sphingomonas y Streptococcus (Chen, Fischbach y Belkaid, 2018).
Según Fariña y col. (2013), Sthaphylococcus es uno de los géneros aislados con mayor frecuencia en estudios de prevalencia de mastitis. Lange y col. (2015) mencionan que las especies pertenecientes al género Streptococcus están vinculadas con la mastitis bovina al ser bacterias oportunistas. Ruiz y col. (2011) en un estudio sobre mastitis subclínica asociada al ordeño manual y mecánico determinan a Staphylococcus sp. como el agente patógeno principal con un 61% de predominancia seguido de StrepDavies y col. (2016) mencionan que el 63% de los eventos potenciales de transmisión y el 38% de incidencia de mastitis clínica es causada por S. uberis en hatos lecheros. Nam y col. (2009) mencionan que cerca del 40% de los casos de mastitis clínica son originados por bacterias Gram (-) vinculadas a la mastitis, y el patógeno no coliforme más comúnmente asociado con las infecciones intramamarias es Sphingomonas sp. como el agente patógeno principal con un 61% de predominancia seguido de Strep tococcus sp. con 29,6% de incidencia. Braga y col. (2018) al realizar una identificación de patógenos de la mastitis bovina por espectrofotometría de masas MALDI-TOF determinaron que aproximadamente el 80% de los casos de mastitis era ocasionado por el grupo de bacterias Staphylococcus aureus, Streptococcus uberis y Streptococcus dysgalactiae.
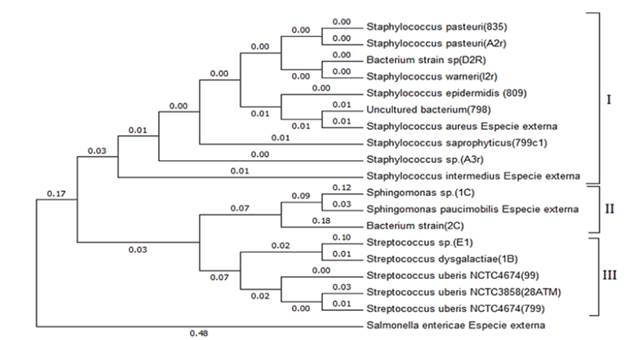
Figura 4 Árbol filogenético elaborado a partir de la secuenciación de la región 16S rRNA de las especies aisladas
Davies y col. (2016) mencionan que el 63% de los eventos potenciales de transmisión y el 38% de incidencia de mastitis clínica es causada por S. uberis en hatos lecheros. Nam y col. (2009) mencionan que cerca del 40% de los casos de mastitis clínica son originados por bacterias Gram (-) vinculadas a la mastitis, y el patógeno no coliforme más comúnmente asociado con las infecciones intramamarias es Sphingomonas sp
3.3 Determinación de resistencia a antibióticos
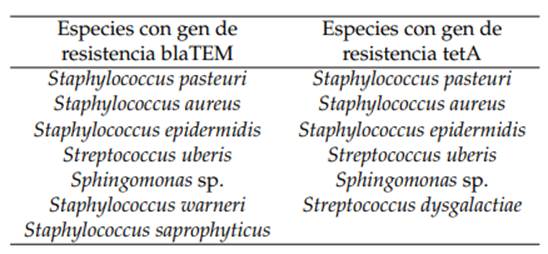
La amplificación del gen de resistencia blaTEM para la familia de los β-lactámicos dio como resultado bandas de aproximadamente 300 pb, datos corroborados por Tao y col. (2014). Se encontró el gen de resistencia en las especies Staphylococcus pasteuri, Staphylococcus warneri, Staphylococcus epidermidis, Staphylococcus saprophyticus, Streptococcus uberis, Sphingomonas sp. y Staphylococcus aureus (Figura 5).
Por otra parte, la amplificación del gen de resistencia tetA para la familia de las tetraciclinas dio como resultado bandas de aproximadamente 250 pb. De acuerdo con Belding y Boopathy (2018), el gen de resistencia para la familia de antibióticos tetraciclinas posee un tamaño de entre 210 a 300 pb. Se encontró el gen de resistencia en las especies Staphylococcus pasteuri, Staphylococcus epidermidis, Streptococcus uberis, Streptococcus dysgalactiae, Staphylococcus aureus y Sphingomonas sp. (Figura 6).

Figura 5 Gel de electroforesis que demuestra la presencia/ausencia del gen de resistencia blaTEM para antibióticos de la familia β-lactámicos.

Figura 6 Gel de electroforesis que demuestra presencia/ausencia de gen de resistencia tetA para antibióticos de la familia de tetracilcinas
Corroborando los resultados obtenidos en es- lactámicos son Staphylococcus warneri, Streptococcus te estudio (Tabla 3), otros estudios de resistencia uberis (Bonetto, 2014), Staphylococcus epidermidis (Faa antibióticos en patógenos relacionados a mastitis riña y col., 2013), Staphylococcus saprophyticus, Sphinhan determinado que las especies resistentes a β- gomonas sp., S. aureus (Ramírez, Fernández y Pala cio, 2018) y Staphylococcus pasteuri (Arrepia, 2008). De igual manera, de acuerdo con estudios realizados por otros autores las especies S. epidermidis presentan resistencia a tetraciclinas (Ramírez, Fernández y Palacio, 2018), S. uberis, S. dysgalactiae, (Florentin, 2007) y S. pasteuri (Savini y col., 2009), mostrando esta última resistencia a numerosas clases de compuestos antibióticos como meticilina/ oxacilina, macrólidos, lincosamidas, estreptograminas y tetraciclinas.
4 Conclusiones
El uso de la técnica molecular permitió identificar género y especie en la mayoría de los microorganismos aislados, con lo cual se concluye que la identificación molecular del agente etiológico es importante en el estudio de la mastitis bovina al ser una prueba más específica que las bioquímicas.
Se pudo determinar que los agentes etiológicos de mayor prevalencia de mastitis bovina en los cantones Cayambe y Pedro Moncayo son los microorganismos pertenecientes a los géneros Staphylococcus y Streptococcus, siendo los más comunes Staphylococcus pasteuri, Staphylococcus warneri, Staphylococcus sp., Staphylococcus epidermidis, Staphylococcus aureus, Staphylococcus saprophyticus, Sphingomonas sp., Streptococcus dysgalactiae, Streptococcu uberis.
La técnica molecular también permitió identificar en las bacterias causantes de la mastitis bovina la presencia de genes de resistencia a dos familias de antibióticos. Las bacterias resistentes a los β-lactámicos fueron Staphylococcus pasteuri, Staphylococcus aureus, Staphylococcus epidermidis, Streptococcus uberis y Sphingomonas sp; y las bacterias determinadas como resistentes a las tetraciclinas fueron Staphylococcus pasteuri, Staphylococcus aureus, Staphylococcus epidermidis, Streptococcus uberis, Sphingomonas sp., Streptococcus dysgalactiae.
La identificación molecular de diferentes géneros y especies bacterianas causantes de la mastitis bovina junto con el estudio de los genes de resistencia a los antibióticos permite tener un diagnóstico más seguro para instaurar un tratamiento eficaz contra esta enfermedad multifactorial que aqueja a las vacas lactantes en la gran mayoría de las unidades productivas lecheras del Ecuador.